Viridiana Candelaria Pérez-Nájera
1 Divizia de biotehnologie, Centrul Universității Ciénega-Universidad de Guadalajara, 47820 Ocotlán, Mexic
Janet Alejandra Gutiérrez-Uribe
2 Tehnologic de Monterrey, Centro de Biotecnología-FEMSA, 64849 Monterrey, Mexic
Marilena Antunes-Ricardo
2 Tehnologic de Monterrey, Centro de Biotecnología-FEMSA, 64849 Monterrey, Mexic
Sergio Hidalgo-Figueroa
3 Cátedra CONACYT, IPICYT/Consorțiu de Investigare, Inovare și Dezvoltare pentru Zonas Áridas, 78216 San Luis Potosi, Mexic
Carmen Lizette Del-Toro-Sanchez
4 Departamentul de Investiții și Construcții în Alimentație, Universitatea din Sonora, 83000 Hermosillo, Mexic
Luis A. Salazar-Olivo
5 Divizia de biologie moleculară, Institutul Potosino pentru Investiții Științifice și Tehnologice (IPICYT), 78216 San Luis Potosi, Mexic
Eugenia Lugo-Cervantes
6 Universitatea Tehnologică Alimentaria, Centrul pentru investiții și asistență în tehnologie și proiectare din statul Jalisco, 44270 Guadalajara, Mexic
Date asociate
Datele utilizate pentru a susține concluziile acestui studiu sunt disponibile de la autorul relevant, la cerere.
Abstract
1. Introducere
Smilax aristolochiifolia Miller (Smilacaceae), cunoscută popular ca zarzaparrilla, este răspândită pe scară largă în Mexic [10] și este utilizată în mod obișnuit ca decocturi de rădăcină indicate ca hipoglicemiante [11] și pentru scăderea în greutate [12]. Cercetările farmacologice au raportat efecte hematopoietice [13], hipoglicemice și hipotensive [14] pentru rădăcina S. aristolochiifolia. Deși potențialul antidiabetic a fost raportat și pentru alte specii Smilax, în principal din S. china [15, 16], identitatea compușilor bioactivi responsabili de efectele antidiabetice ale S. aristolochiifolia, precum și mecanismele lor de acțiune sunt încă necunoscute. Prin urmare, ne propunem să identificăm compușii bioactivi majori din rădăcina S. aristolochiifolia și să caracterizăm efectele acestora asupra activităților enzimatice ale α-amilazei și α-glucozidazei.
2. Materiale și metode
2.1. Materiale
Plantele de Smilax aristolochiifolia Miller (inclusiv rădăcinile) au fost colectate în Apazapan, Veracruz, Mexic (19 ° 19′25.6 ″ N și 96 ° 43′17.3 ″ W) în octombrie 2015. Materialul vegetal a fost autentificat de Dr. M. Chazaro (Departamentul de Biologie, Universitatea Veracruzana), și un specimen de voucher (10855) au fost depuse în Institutul de ecologie ierbar (IE-XAL), Xalapa, Veracruz, Mexic. α-glucozidază (EC 3.2.1.20, de la Saccharomyces cerevisiae, 28 U/mg), acarboză, ρ-nitrofenil-α-D-glucopiranozidă (pNPG), α-amilază pancreatică porcină (EC 3.2.1.1, tip VI-B, din pancreasul porcin, ≥10 U/mg) și reactiv de acid 3,5-dinitrosalicilic (DNS) au fost achiziționați de la Sigma-Aldrich Co. (St. Louis, MO, SUA). Amidonul solubil a fost achiziționat de la Jalmek Científica (Monterrey, NL, Mexic).
2.2. Prepararea extractului de rădăcină S. aristolochiifolia
Rădăcina plantei a fost uscată la întuneric la temperatura camerei și materialul uscat a fost apoi măcinat cu o moară cu bile. Testele preliminare au arătat că extracția rădăcinilor de S. aristolochiifolia prin infuzie apoasă sau macerare hidroetanolică dă naștere la același profil de eluare (Figura S1), deși macerarea a produs un randament de 2 ori mai mare decât infuzia (15,28% prin infuzie și 30,11% prin macerare ). Extracția a fost efectuată prin macerare la temperatura camerei (25 ° C) și agitarea peste noapte folosind un raport solid: lichid de 1: 20 g/v în etanol: apă (1: 1, v/v) ca solvent. Extractul de rădăcină S. aristolochiifolia (SAR) a fost obținut prin filtrare pe hârtia Whatman nr. 4. Apoi, etanolul a fost eliminat prin concentrație sub vid (IKA RV 10 digital, Staufen, Germania) la 40 ° C și apă prin liofilizare. SAR uscat a fost depozitat la -80 ° C până la utilizare.
2.3. Cromatografie rapidă de partiție centrifugă
Fracțiunile din SAR au fost obținute într-un instrument preparativ de cromatografie rapidă de partiție centrifugă (FCPC) (Kromaton, Angers, Franța), cu o capacitate a rotorului de 1 L, acționat în mod dual: 0-57 min în modul descendent și 58-120 min în modul ascendent la 1000 rpm și un debit de 10 mL/min folosind acetat de etil: apă (1: 1 v/v) ca sistem de solvent cu două faze, conform testelor preliminare (Tabelul S1). SAR (10 g) a fost dizolvat în 160 ml de sistem de solvent, filtrat și pompat în rotor. O sută douăzeci de fracții au fost colectate și grupate în grupuri de 10 fracții în funcție de similaritatea valorilor coeficientului de partiție (kd) pentru a facilita analiza lor. Un total de 12 bazine au fost concentrate până la uscare la 45 ° C sub presiune redusă (EZ-2 Plus, Genevac Ltd., Marea Britanie) și depozitate la -20 ° C până la testare.
2.4. Analiza cromatografiei lichide de înaltă performanță
SAR și fracțiile sale derivate din FCPC au fost analizate prin HPLC-DAD (Agilent Technologies, seria 1200, Santa Clara, CA) conform metodei descrise de Becerra-Moreno și colab. [17] cu unele modificări. Compușii au fost separați într-o coloană Luna 5 U C18, 4,6 mm ID × 250 mm (5 μm) (Phenomex, Torrance, CA). Faza mobilă a fost constituită din solvent A, apă de calitate HPLC (BDH, Poole, Marea Britanie) acidificată cu acid formic 0,1% (CTR Scientific, Monterrey, NL, Mexic) și solvent B, metanol de calitate HPLC (BDH, Poole, Marea Britanie), folosind un gradient la un debit de 0,8 ml/min. Proporția fazei mobile a fost menținută după cum urmează: 0-3 min (B, 0% până la 18%); 3-8 min (B, 18% până la 30%); 8-35 min (B, 30% până la 42%); 35-40 min (B, 42% până la 48%); 40-45 min (B, 48% până la 60%); 45-50 min (B, 60% până la 100%); 50-60 min (B, 100% până la 0%). S-au obținut cromatograme la 280 nm, s-au injectat 10 μl de probă și s-au colectat spectre de absorbție UV. Rezultatele cuantificării au fost exprimate ca echivalenți de acid clorogenic sau caempferol-3-O-glucozid, pe baza curbei de calibrare a standardelor corespunzătoare.
Identificarea compușilor majori a fost efectuată prin cromatografie lichidă cuplată cu spectrometrie de masă timp de zbor (LC/MS-TOF) (seria 1100, Agilent Technologies, Santa Clara, CA), utilizând aceleași condiții cromatografice descrise mai sus. Ionizarea a fost efectuată utilizând o sursă de ionizare cu electrospray în modul pozitiv (ESI +) cu următoarele condiții: interval pentru scanarea în masă acoperit de la m/z 140 la 1000, temperatura azotului gazului setată la 350 ° C, debitul gazului la 11 L/min, presiunea nebulizatorului la 50 psi, 3500 V tensiune capilară și 50 V în fragmentor. Cromatogramele ionice extrase au fost obținute luând în considerare masa exactă a compusului utilizând software-ul Analyst QS 1.1 (Applied Biosystems, Carlsbad, CA).
2.5. Testele de inhibare a enzimelor și studiul mecanismului de acțiune
2.5.1. Testarea activității α-amilazei
Activitatea inhibitoare a α-amilazei a SAR, CAF și ABF a fost determinată prin măsurarea puterii de reducere a oligozaharidei eliberate din amidonul solubil conform metodei Miller [18]. O serie de teste la concentrații variate atât de substrat cât și de inhibitor au fost efectuate pentru a determina tipurile de inhibiție. SAR, CAF și ABF au fost preparate la concentrații de 1 până la 200 μg/ml în tampon fosfat 20 mM cu 6,2 mM clorură de sodiu la pH 6,9. A-amilaza pancreatică porcină la 5 U/mL și soluții solubile de amidon 1% au fost preparate în același tampon. Toate soluțiile au fost preparate imediat înainte de fiecare test.
Alicote ale unei soluții de α-amilază de 500 μL și a unei soluții de probă de 500 μL au fost amestecate într-un tub Eppendorf de 15 ml și incubate la 20 ° C timp de 10 min. Reacția a fost inițiată prin adăugarea de 1 ml soluție de amidon în amestec și incubată timp de 10 minute la 20 ° C. Apoi, s-a adăugat 1 ml soluție de reactiv de acid 3,5-dinitrosalicilic (DNS) urmat de încălzire în apă clocotită timp de 15 minute pentru a dezvolta culoarea. Reacția a fost oprită prin răcirea în apă cu gheață. Amestecul de reacție a fost diluat cu 9 ml de apă distilată și absorbanța a fost citită la 540 nm folosind un spectrofotometru (UV-VIS 6405, JENWAY, UK). Acarboză a fost utilizată ca martor pozitiv și tampon fosfat ca martor negativ. Procentul de inhibare a fost calculat în urma ecuației:
2.5.2. Testarea activității α-glucozidazei
Activitatea inhibitoare a α-glucozidazei SAR, CAF și ABF a fost testată prin metoda pNPG (ρ-nitrofenil-α-D-glucopiranozidă) [19]. Pe scurt, amestecurile de reacție constând din 25 μL de α-glucozidază de la S. cerevisiae (0,2 U/mL) (Sigma-Aldrich, G5003) și 25 μL de concentrații diferite (1 până la 200 μg/mL) de soluții de probă au fost preincubate într-un Placă cu 96 de godeuri la 20 ° C timp de 10 min. Apoi, reacția a fost începută prin adăugarea a 50 μL de 2 mM pNPG în fiecare godeu. După 20 min, reacția a fost oprită prin adăugarea a 50 μL de 0,2 M Na2CO3. Toate soluțiile au fost preparate imediat înainte de fiecare test și 20 mM tampon fosfat cu 6,2 mM clorură de sodiu la pH 6,9 a fost utilizat ca vehicul. Produsul ρ-nitrofenol eliberat din substratul pNPG a fost utilizat pentru a cuantifica activitatea enzimatică; absorbanța a fost măsurată la 405 nm într-un cititor de microplăci (modelul Bio-Rad 550, Berkeley, CA). Procentul de inhibiție a fost calculat conform (1).
2.5.3. Determinarea modelului inhibitor enzimatic prin analiza cinetică
Tipul de inhibare a enzimei a fost determinat grafic folosind graficul Lineweaver - Burk. Activitatea de inhibare (IC50) a fost utilizată pentru a evalua eficacitatea unui inhibitor. Valoarea IC50 este definită ca concentrația unei substanțe testate necesară pentru a obține inhibarea la jumătate maximă a unei reacții date. Valorile IC50 au fost calculate utilizând regresia neliniară și funcția logistică.
2.6. Modelarea omologică a α-glucozidazei
Spre deosebire de α-amilaza, structura cristalografică pentru enzima α-glucozidază nu este disponibilă; prin urmare, modelul 3D al α-glucozidazei din Saccharomyces cerevisiae a fost generat pe baza similitudinii secvenței prin utilizarea modelării omologice. Secvența de aminoacizi a proteinei țintă a fost preluată de la NCBI (https://www.ncbi.nlm.nih.gov/) cu ID:> P53341.1. Serverul BLASTp a fost utilizat împotriva bazei de date Protein Data Bank pentru a găsi șablonul de structură adecvat pentru modelul de omologie. Alinierea dintre secvențe a fost realizată folosind programul MODELLER v.9.18. Au fost construite o sută de modele, iar modelul unic a fost selectat prin scorul DOPE (Discrete Optimized Protein Energy). Modelul final a fost validat folosind două instrumente ProSa (Protein Structure Analysis) și QMEAN (Qualitative Model Energy Analysis).
2.7. Studii de andocare moleculară
Studiile de andocare moleculară au fost utilizate pentru a explora modul de legare dintre ligand și receptor [20]. Conform rezultatelor testelor enzimatice și tipului de inhibare, am investigat modurile de legare ale acidului clorogenic și astilbin împotriva enzimelor α-amilază și α-glucozidază. Mai întâi, structura tridimensională a a-amilazei pancreatice porcine cu malto-oligozaharide (PDB ID: 1UA3) a fost obținută din baza de date Protein Data Bank (PDB). Pentru calcul, malto-oligozaharidele au fost conservate și toate moleculele de apă au fost îndepărtate din structura cristalografică. Andocarea moleculară a fost efectuată folosind AutoDock 4.2 și AutoDock Tools (ADT, v.1.5.6). Dimensiunile grilei au fost ajustate la 60 × 60 × 60 puncte separate de 0,375 Å. PyMOL (PyMOL Molecular Graphics System, San Carlos, CA, SUA) și Discovery Studio Visualizer v.17.2.0.16349 (BIOVIA, San Diego, CA, SUA) au fost utilizate pentru vizualizare.
Pe de altă parte, structurile 3D ale acidului clorogenic și astilbin au fost obținute de la PubChem în baza de date NCBI. Dimensiunile grilei, care reprezintă coordonatele parametrilor în care ligandul poate fi mutat, au fost 30 × 40 × 40 puncte separate de 1,0 Å. Pentru stabilirea grilei, a fost ales pe baza siturilor de legare raportate în inhibiția necompetitivă pentru α-glucozidază, acoperind reziduurile ASP214, GLU276 și ASP349, ILE149, PRO150 și ASP232 și SER311, PRO312, VAL319, THR310, GLY309, VAL308, ASP307, PHE321 și PRO320 [21-23]. În parametrii impliciți, algoritmul genetic Lamarckian (LGA) a fost ales pentru calcule de andocare și s-au efectuat 100 de experimente per ligand. Comparativ cu rigiditatea proteinei, ligandul a rămas flexibil. În rezultatele andocării moleculare, poziția cu cea mai mică energie de andocare și numărul maxim de conformații a fost selectată pentru a reprezenta cel mai favorabil mod de legare prevăzut de acest program.
2.8. Analize statistice
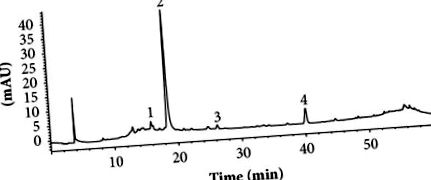
Testele enzimatice au fost efectuate în triplicat. Rezultatele au fost analizate folosind Statgraphics Centurion XVII v.17.2.00 cu testul HSD al lui Tukey. Pentru fiecare set de date, P Figura 1) a prezentat doi constituenți fenolici principali, vârful 2 eluând la 18,59 min și vârful 4 eluând la 40,37 min. Doi constituenți minori SAR, vârfurile 1 și 3, nu au fost luați în considerare în lucrarea de față. Vârfurile 2 și 4 au fost identificate provizoriu în funcție de absorbția UV, m/z, modelele de fragmentare și datele raportate anterior. Spectrul UV-vis al vârfului 2 a prezentat benzi caracteristice ale unui reziduu de cofeină cu λmax de 240 și 327 nm (Figura 2 (b)) și un ion molecular de 355,09 m/z [M + H +] (Figura 2 (a) ) corespunzând acidului clorogenic (Figura 2 (c)). Identitatea acidului clorogenic a fost confirmată de timpul de retenție standard (datele nu sunt prezentate). Spectrul UV-vis al vârfului 4 a avut o absorbție maximă de 290 nm (Figura 2 (e)) și un ion molecular de 451,12 m/z [M + H +] (Figura 2 (d)), care este caracteristic astilbinului, un compus flavonoid (Figura 2 (f)) [24]. Rezultatele noastre constituie primul raport al prezenței acidului clorogenic și a astilbinului în S. aristolochiifolia, deși ambii compuși au fost raportați anterior pentru alte specii din genul Smilax [25-30].
HPLC-UV/Vis cromatogramă prezentată la 280 nm de extract hidroetanolic de rădăcină S. aristolochiifolia. Condiții: coloană C18 cu fază inversă (4,6 × 150 mm, 5 μm, Phenomex); faza mobilă, apă acidulată cu 0,1% acid formic și metanol folosind un gradient de concentrație (vezi metodologia); debit: 0,8 ml/min.
- Dieta cu extract de boabe de cafea verde pur 800 50% acid clorogenic Groupon
- Extract pur de forskolin pentru pierderea în greutate 300 mg cu 40% extract standardizat de rădăcină Coleus Forskohlii
- Informații despre medicamentul acidului nicotinic (niacină; vitamina B3) myVMC
- Extract pur de boabe de cafea verde prezentat pe Dr Oz - Forum
- Beneficiile rădăcinii bujorului