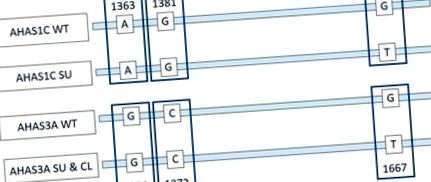
Relația dintre AHAS1C și AHAS3A și pozițiile unei mutații cheie. Reprezentarea catenelor de codificare ale genei AHAS1C și genei AHAS3A în tipul sălbatic (WT), genom editat pentru sulfoniluree și toleranță la imidazolinonă (SU) și soiuri de canola Clearfield (CL). Aceste regiuni ale genelor AHAS1C și AHAS3A sunt identice, cu excepția bazelor indicate. Diferențele în numerotarea celor două gene se datorează diferențelor în secvențe în amonte de regiunea descrisă. Cele două SNV la 1363/1354 și la 1381/1372 diferențiază AHAS1C de AHAS3A. SNV la 1676/1667 diferențiază AHAS1C SU, AHAS3A SU și AHAS3A CL de AHAS1C WT și AHAS3A WT. Vezi lucrările citate în această secțiune pentru detalii despre structura genei AHAS.
Porțiune din secvențierea Sanger a segmentului genei AHAS1C care se întinde pe locul editării genomului, la poziția 1676, evidențiat în albastru. Această bază este homozigotă G (tip sălbatic) în canola Clearfield soiul 5545 CL, homozigotă T (mutantă) în SU canola soiul 40K și G/T heterozigotă în varietățile de canola SU C5507 și C1511. Regiunea secvențiată s-a extins de la SNV-uri specifice AHAS1C la pozițiile 1363 și 1381 până la poziția 1704, verificând locația editării genomului la poziția 1676. Locația exemplului folosit pentru secvențierea este indicată în Figura 3 .
Secvența unei părți a exonului 2 al genei SU canola AHAS1C care prezintă alinieri ale primerilor și sondei pentru ampliconul de 334 bp. Exemplul direct și sonda corespund secvenței țintă în timp ce exemplul invers este complementar secvenței țintă. Steaua din exemplul direct indică G care este caracteristic genei AHAS 1C, distingându-l de gena AHAS 3A care are un C în acel loc. Steaua din exemplul invers indică SNV unic care conferă toleranță la erbicide pentru AHAS 1C SU și AHAS 3A SU și CL. Exemplul de secvențiere este utilizarea oligonucleotidelor ca exemplu pentru secvențierea Sanger prezentată în Figura 2 .
Curbele cantitative de amplificare PCR pentru rezultatele prezentate în Tabelul 2, cu primeri specifici pentru gena CruA (Panoul A) sau cu primeri specifici genei SU canola AHAS1C-SU (Panoul B). În panoul B, amplificarea a avut loc numai cu soiurile de canola SU 40K, C1511 și C5507, în timp ce în panoul A, amplificarea a avut loc și cu soiurile de canola Clearfield.
Curbele cantitative de amplificare PCR pentru rezultatele prezentate în Tabelul 3. Panoul A, amplificare utilizând metoda qPCR specifică pentru gena de referință endogenă a canola (CruA); Panoul B, amplificare utilizând metoda qPCR specifică pentru AHAS1C-SU SNV a canolei SU. Controalele pozitive sunt ADN-ul de la canola SU 40K la 300 (a), 30 (b) și 3 (c) ng 40K ADN per reacție.
Curbele tipice de amplificare pentru rezultatele prezentate în Tabelul 3 din SU canola ADN varietate 40K amestecat la cinci concentrații cu Clearfield canola ADN varietate 5545 CL.
Abstract
Relația dintre AHAS1C și AHAS3A și pozițiile unei mutații cheie. Reprezentarea catenelor de codificare ale genei AHAS1C și genei AHAS3A în tipul sălbatic (WT), genom editat pentru sulfoniluree și toleranță la imidazolinonă (SU) și soiuri de canola Clearfield (CL). Aceste regiuni ale genelor AHAS1C și AHAS3A sunt identice, cu excepția bazelor indicate. Diferențele de numerotare ale celor două gene se datorează diferențelor în secvențele din amonte de regiunea descrisă. Cele două SNV-uri la 1363/1354 și la 1381/1372 diferențiază AHAS1C de AHAS3A. SNV la 1676/1667 diferențiază AHAS1C SU, AHAS3A SU și AHAS3A CL de AHAS1C WT și AHAS3A WT. Vezi lucrările citate în această secțiune pentru detalii despre structura genei AHAS.
Porțiune din secvențierea Sanger a segmentului genei AHAS1C care se întinde pe locul editării genomului, la poziția 1676, evidențiat în albastru. Această bază este homozigotă G (tip sălbatic) în canola Clearfield soiul 5545 CL, homozigotă T (mutantă) în SU canola soiul 40K și G/T heterozigotă în varietățile de canola SU C5507 și C1511. Regiunea secvențiată s-a extins de la SNV specifice AHAS1C la pozițiile 1363 și 1381 până la poziția 1704, verificând locația editării genomului la poziția 1676. Locația exemplului utilizat pentru secvențierea este indicată în Figura 3 .
Secvența unei părți a exonului 2 al genei SU canola AHAS1C care prezintă alinieri ale primerilor și sondei pentru ampliconul de 334 bp. Exemplul direct și sonda corespund secvenței țintă în timp ce exemplul invers este complementar secvenței țintă. Steaua din exemplul direct indică G care este caracteristic genei AHAS 1C, distingându-l de gena AHAS 3A care are un C în acel loc. Steaua din exemplul invers indică SNV unic care conferă toleranță la erbicide pentru AHAS 1C SU și AHAS 3A SU și CL. Exemplul de secvențiere este utilizarea oligonucleotidelor ca exemplu pentru secvențierea Sanger prezentată în Figura 2 .
Curbele cantitative de amplificare PCR pentru rezultatele prezentate în Tabelul 2, cu primeri specifici pentru gena CruA (Panoul A) sau cu primeri specifici pentru gena SU canola AHAS1C-SU (Panoul B). În panoul B, amplificarea a avut loc numai cu soiurile de canola SU 40K, C1511 și C5507, în timp ce în panoul A, amplificarea a avut loc și cu soiurile de canola Clearfield.
Curbele cantitative de amplificare PCR pentru rezultatele prezentate în Tabelul 3. Panoul A, amplificare utilizând metoda qPCR specifică pentru gena de referință endogenă a canola (CruA); Panoul B, amplificare utilizând metoda qPCR specifică pentru AHAS1C-SU SNV a canolei SU. Controalele pozitive sunt ADN-ul de la canola SU 40K la 300 (a), 30 (b) și 3 (c) ng 40K ADN per reacție.
Curbele tipice de amplificare pentru rezultatele prezentate în Tabelul 3 din SU canola ADN varietate 40K amestecat la cinci concentrații cu Clearfield canola ADN varietate 5545 CL.
- Alimente Iradierea UV-C fără text integral a fileurilor laminate de șuncă inoculate cu Yersinia
- Sambucus nigra L
- Alimente modificate genetic Eseu dăunător sau util - Eseuri argumentative gratuite pentru studenți
- Gene Gag domesticite cu text complet gratuit Gena de Drosophila LTR Retrotranspozonii este implicată în
- Geosciences Free Full-Text Înțelegerea sistemului Permafrost - Hydrate și a metanului asociat