Date asociate
Abstract
1. Introducere
Obezitatea a fost recunoscută ca o problemă de sănătate publică de vârf în întreaga lume [1]. Aproximativ două treimi din SUA sunt supraponderali (indicele de masă corporală (IMC) ≥ 25 kg/m2) și peste 50% dintre acești supraponderali sunt obezi (IMC ≥ 30 kg/m2) [2]. Un număr tot mai mare de studii cu cohorte mari au demonstrat că obezitatea este susceptibilă la un risc de boli, în special de cancer [1,2,3]. Studiile epidemiologice la scară largă au arătat, de asemenea, că femeile obeze au o incidență mai mare de a dezvolta cancer de sân, iar pacienții cu cancer de sân obezi tind să aibă un prognostic slab [4,5,6,7,8]. S-au descoperit că mai multe mecanisme explică corelația dintre obezitate și cancerul de sân, cum ar fi modificările induse de obezitate ale funcțiilor endocrine adipoase, imunitatea sistemică și homeostazia metabolică [9,10,11,12,13]. Cu toate acestea, mecanismele exacte care stau la baza progresiei cancerului de sân mediat de obezitate rămân incomplete.
2. Rezultate
2.1. Dietele bogate în grăsimi induc obezitatea în Brca1 -/-; p53 +/− Șoareci pentru analiza scARN-seq
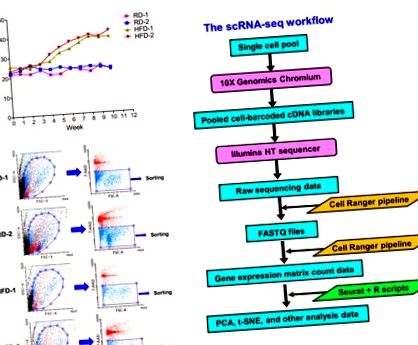
Pentru a induce Brca1 -/-; P53 +/− șoareci pentru a dezvolta obezitate, am hrănit șoareci femele de două luni cu diete bogate în grăsimi (HFD) timp de 10 săptămâni. Șoarecii femele cu vârsta similară în grupul cu greutate normală au fost hrăniți cu diete regulate (RD) pentru aceeași perioadă de timp. În timpul perioadei de hrănire a dietei, le-am măsurat greutățile săptămânal. Figura 1 A arată că șoarecii hrăniți cu diete bogate în grăsimi au crescut semnificativ în greutate până la 40,11 ± 2,14 g în medie după 10 săptămâni, comparativ cu grupul de șoareci martor cu greutate medie de 22,33 ± 0,52 g. La sfârșitul experimentelor de hrănire a dietei, șoarecii experimentali au fost eutanasiați și glandele lor mamare au fost recoltate pentru prepararea unei singure celule, așa cum este descris în „Materiale și metode”. În acest studiu, ne-am concentrat doar asupra impactului obezității induse de HFD asupra fibroblastelor stromale și a celulelor imune. Pentru a îmbogăți aceste tipuri de celule, am folosit anticorpul anti-EpCAM pentru a elimina celulele epiteliale mamare. Fracțiile de celule non-epiteliale îmbogățite au fost colorate cu colorant 7-AAD și apoi supuse sortării celulelor pentru a izola celule unice viabile (Figura 1 B). Având în vedere că celulele moarte au fost colorate cu 7-AAD, celulele 7-AAD-negative au fost închise pentru sortarea celulelor pentru a purifica celule unice viabile (Figura 1 B). Celulele unice viabile sortate au fost supuse analizei scARN-seq folosind sistemul 10x Genomics Chromium.
Analiza scARN-seq a sortării celulelor activate de fluorescență (FACS) - celule sortate izolate din glandele mamare de HFD sau RD Brca1 -/-; p53 +/− șoareci. (A) Hrănirea cu diete bogate în grăsimi a indus obezitatea în Brca1 -/-; p53 +/− șoareci. Datele privind greutatea corporală a șoarecilor experimentali au fost reprezentate grafic în raport cu informațiile săptămânale. Patru femei C57BL6 Brca1 -/-; Șoarecii p53 +/− au fost implicați în experiment (grupul cu dietă obișnuită: RD-1 și RD-2; grupul cu dietă bogată în grăsimi: HFD-1 și HFD-2). (B) Sortarea FACS a celulelor mamare viabile. Celulele non-epiteliale îmbogățite au fost colorate cu colorant 7-AAD și apoi sortate folosind condiția închisă prezentată în datele FACS. 7-AAD-celule negative au fost închise pentru sortarea celulelor viabile. (C) Diagrama fluxului de lucru scRNA-seq.
Așa cum se arată în Figura 1 C, proiectarea tehnologică a sistemului 10 × Genomics Chromium pentru scARN-seq este microfluidică pe bază de picături cu microcanale pentru generarea picăturilor de emulsie apoasă-ulei [30]. Fiecare picătură este compusă în mod ideal din reactivul pentru transcriere inversă (RT), o singură celulă și o margelă conjugată cu oligonucleotide. Secvența oligonucleotidică are secvența adaptor universal concepută pentru amplificarea PCR, secvența codului de bare celulară specifică fiecărei mărgele, secvența identificatorului molecular unic (UMI) pentru secvențierea analizei citirii și secvența oligo (dT) pentru RT [30]. Procesul de transcriere inversă are loc în picături încapsulate și ADNc-urile lor sintetizate sunt reunite, amplificate prin PCR și apoi supuse secvențierii de generație următoare.
2.2. Analiza scARN-Seq identifică diferite subtipuri moleculare de fibroblaste stromale mamare în Brca1 -/-; p53 +/− șoareci
Datele brute citite secvențierea ARN cu o singură celulă au fost procesate prin conducta de analiză Cell Ranger pentru a genera rapoarte QC scRNA-seq (Figurile S1 - S4) și trei seturi de date brute (cod de bare de celule, caracteristică genică și matrice de numărare) pentru fiecare probă de celule C) [ 31]. După cum se arată în rapoartele QC, peste 600 de celule pe probă (RD-1: 1138, RD-2: 686, HFD-1: 1165, HFD-2: 903) au fost secvențiate cu succes (Figurile S1 - S4). Mediile totalului de gene detectate și gene mediane pe celulă pentru aceste patru probe de celule sunt de 15.553 ± 309 și respectiv 1492 ± 257. Procentul mediu de citiri mapate cu încredere la transcriptomul mouse-ului este de 66,05% ± 7,69%. Am efectuat o analiză scRNA-seq a acestor trei seturi de date (cod de bare celular, caracteristică genică și matrice de numărare) pentru fiecare probă de celule folosind instrumentul de bioconductori bazat pe R numit Seurat (Figura 1 C) [32,33]. Deoarece au existat două seturi de date scARN-seq pentru fiecare grup de dietă, am exploatat Seurat pentru a le integra împreună înainte de analiza reducerii dimensionalității [33]. Prin analiza utilizând algoritmi de reducere a dimensionalității construiți în Seurat, s-au generat graficele de analiză a componentelor principale (PCA; Figura 2 A) și încorporarea stochastică a vecinilor (t-SNE; Figura 2 B) pentru a clasifica tipurile de celule (Figura 2). Graficele t-SNE pentru grupurile HFD și RD prezintă 14 clustere de celule diferite (Figura 2 B), indicând prezența a cel puțin 14 tipuri de celule distincte în probe de celule sortate.
Reducerea dimensionalității și analizele de căldură ale datelor secvențiale ARN cu o singură celulă (scRNA) -seq. (A) Analiza grafică PCA a datelor scARN-seq. (B) Analiza grafică t-SNE a datelor scARN-seq. Numele de tip celular sunt adnotate în grafice pe baza genelor marker identificate specifice tipului de celulă. (C) Harta de căldură a genelor de vârf exprimate diferențial în fiecare subtip de fibroblaste stromale (Top 12 gene pentru T1 - T4 și top 6 gene pentru T5).
Prin analiza grupului de celule individuale utilizând algoritmul de identificare a markerilor genici construit în Seurat, am identificat markeri genici specifici tipului de celulă pentru clasificarea tipului de celule. Expresia markerilor genetici identificați în aceste 14 grupuri de celule a fost afișată în comploturi de vioară (Vlnplots; Figura 3) și comploturi genetice t-SNE (Figura 4). Pe baza acestor markeri genici specifici tipului de celulă identificați arătați în Figura 3 și Figura 4, am putut atribui nume de tipuri de celule cunoscute la 13 din 14 grupuri de celule (Figura 2 B), inclusiv fibroblaste stromale (Vim +; 5 diferite clustere de celule), celule T de memorie (Cd3d +, Lef1 +, Crem +), celule T naive (Cd3d +, Lef1 +, Sell +), celule B (Cd79a +), celule NK (Gzma +, Nkg7 +), monțiți (Cd14 +, Tnip3 +, Retnla +, Cd68 +), macrofage (Cd14 +, Retnla +, Cd68 +, Cd163 +, C1qb +), celule dendritice dermice (DC dermice; Cd14 +, Clec4e +) [34] și dendritice celule (DC; Siglech +) [35]. Prin analiza scARN-seq, am identificat pentru prima dată cinci tipuri diferite de fibroblaste stromale prezente în Brca1 -/-; p53 +/− glande mamare, incluzând stroma T1 (Tnfaip6 înaltă), stroma T2 (Igfbp5 +), stroma T3 (iunie înaltă), stroma T4 (Rgcc +) și stroma T5 (Klf2 înaltă) (Figura 2, Figura 3 și Figura 4).
Analiza expresiei genelor marker specifice tipului de celulă pe baza datelor scARN-seq. Expresia acestor gene marker a fost prezentată în Vlnplots. Algoritmul de analiză diferențială a expresiei genelor încorporat în pachetul Seurat a fost utilizat pentru a identifica genele marker exprimate predominant sau unic în fiecare tip de celulă.
- A nimerit-o! Steaua Jacques Torres dezvăluie 60 de kilograme
- Naomi Campbell dezvăluie că mănâncă o singură masă pe zi, PerthNow
- Natalie Dormer dezvăluie abs impresionante asupra femeilor; Acoperire de sănătate
- Luna Națională a Nutriției Cum să urmezi o dietă îmbogățită cu super-alimente, expertul dezvăluie - NDTV Food
- Diferențele de rasă în calitatea dietei pentru negrii și albii cu insecuri alimentare urbane dezvăluie rezistența la