Svetlana Lebedeva
Institutul de Biologie Moleculară, Mainz, Germania
António M. de Jesus Domingues
Institutul de Biologie Moleculară, Mainz, Germania
Falk Butter
Institutul de Biologie Moleculară, Mainz, Germania
René F. Ketting
Institutul de Biologie Moleculară, Mainz, Germania
Date asociate
ABSTRACT
Proteina de legare a ARN-ului FUS este implicată în transcriere, îmbinarea alternativă a genelor neuronale și repararea ADN-ului. Mutațiile din FUS au fost legate de boli neurodegenerative umane, cum ar fi SLA (scleroză laterală amiotrofică). Am întrerupt genetic fusul din zebră (Danio rerio) folosind sistemul CRISPR-Cas9. Animalele knockout fus sunt fertile și nu au prezentat niciun fenotip distinctiv. Mutația fusului induce modificări ușoare în expresia genelor la nivelul transcriptomului și proteomei din creierul adult. Am observat o influență semnificativă a fondului genetic asupra expresiei genelor și a utilizării 3'UTR, care ar putea masca efectele pierderii Fus. Spre deosebire de morfanții fus publicați, mutanții fizi zigotici materni nu prezintă degenerescență motoneuronală și prezintă activitate locomotorie normală.
Abrevieri
Introducere
Rezultate
Fus alele knockout în pești zebră
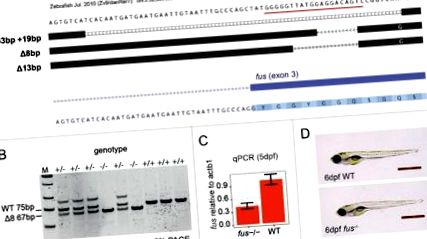
Am folosit tehnologia CRISPR-Cas9 24 pentru a întrerupe gena fus zebrafish cu un singur ARN ghid care vizează exonul 3 (Fig. 1A). Toate analizele ulterioare au fost efectuate pe alela care șterge 8 perechi de baze, rezultând un schimb de cadre și un codon de oprire prematur (alela „Δ8”) (Fig. 1A, B). ARNm de 8 fuz era încă prezent la animalele eliminabile, deși era de aproximativ 2 ori mai puțin abundent decât ARNm în greutate (Fig. 1C). Anticorpii publicați folosiți pentru a detecta proteina Fus din peștele zebră pe Western blot 21 nu au reușit să detecteze Fus în embrionii de tip sălbatic din mâinile noastre (neprezentat). Prin urmare, am folosit spectrometria de masă (MS) pentru a determina dacă ARNm de fus8 fus era încă capabil să producă proteine. Pentru a minimiza o posibilă contribuție de proteine depuse matern, am analizat embrioni de 5 zile dintr-un incruciș heterozigot. Nu au fost detectate peptide unice proteinei Fus în niciunul dintre cele 4 replicate ale embrionilor fus -/-, în timp ce 2 peptide unice Fus au fost prezente în ¾ replicatele fraților de tip sălbatic (Tabelul 1). Această analiză ne-a condus la concluzia că proteina Fus nu a fost produsă și alela fus Δ8 este o mutație a pierderii funcției.
Generarea și validarea peștilor zebră fus knockout. (A) alele fuzibile generate de CRISPR-Cas9. O captură de ecran din browserul genomului UCSC prezintă secvențe aliniate de la animale F1 heterozigoți. Site-ul țintă CRISPR din fusul exon 3 este subliniat cu roșu. (B) Un exemplu de gel PAGE pentru genotiparea alelei Δ8. M = Marker cu greutate moleculară mică. Produsele de tip sălbatic (75bp) și fus -/- (67bp) pot fi discriminate. (C) Cuantificarea ARNm fus în raport cu ARNm WT la embrioni 5dpf. (D) Morfologia normală a WT și a embrionului fus -/- la 6 d post fertilizare (dpf). Bara de măsurare este de 1 mm.
tabelul 1.
Identificarea Fusului prin spectrometrie de masă (MS). Peptidele unice pentru proteina Fus au fost identificate numai în analiza SM a embrionilor WT 5dpf și a creierului adult, dar nu și în eliminarea Fus. Cross (x) înseamnă că peptida a fost identificată în proba respectivă. Mai jos: secvența proteică completă Fus, peptidele detectate unice pentru Fus sunt îndrăznețe și subliniate.
| Genul mai sălbatic | picior -/- | ||||||||
| Probă | Secvenţă | repl. 1 | repl. 2 | repl. 3 | repl. 4 | repl. 1 | repl. 2 | repl. 3 | repl. 4 |
| Embrion | AAIDWFDGKDFNGNPIK | X | |||||||
| TGLPMINLYTDR | X | X | |||||||
| Creier | AAIDWFDGKDFNGNPIK | X | X | X | X | ||||
| CSNPSCGNLNFSWR | X | X | X | ||||||
| TGLPMINLYTDR | X | ||||||||
MASNDYGQTSSHGYGGYGGQSGQSYSQPSAQNYSQQSYGGYNQSSESSSAPYNQGGYSSNYGQSQSGGYGSQAPSQGYSQSSQSYSSGGYSNTSQPPPAQSGGYSQQSSYSGYNQSSPASAPGGYSSSSQSSGYGQQQQQSGGGYGGSGGQSGGYGSSGGQSSGFGGSGGQHQSSQSGGGSYSPSPNYSSPPPQSYGQQSQYGQGGYNQDSPPMSGGGGGGGYGGQDGGYSQDGRGGRGRGGGFGGRGAGGFDRGGRGGPRGRGGMGMGDRGGFNKFGGPRDHGAGGPNMQEQDNSDNNTIFVQGLGDDYTVDSVADYFKQIGIIKVNKKTGLPMINLYTDRETGKLKGEATVSFDDPPSAKAAIDWFDGKDFNGNPIKVSFATRRAEFGRGGSSGGMRGGRGRGGPMGRGGFGGGGGGGGGGGGFQGNNGGGSGNGGGQQRAGDWKCSNPSCGNLNFSWRNECNQCKEPKPEGSGGGMSPMGGGFGGERGRSGFDRGGFRGRGGDRGGFRGGRGGDRGGFGPGKMDSRGDHRHDRRDRPY
Analiza transcriptomului și proteomului creierului knockout fus
Transcriptom și proteom al creierului fus -/-. (A) Adult. creierele de pește zebră de la frații fus -/- sau WT au fost utilizate pentru a cuantifica simultan ARNm prin secvențierea ARN și proteinele prin spectrometrie de masă. În total, s-au măsurat transcriptomii de 5 fuz -/- și 4 WT și proteomi de 4 fus -/- și 4 animale WT. (B) Diagrama vulcanică a modificărilor de pliere a ARNm afișează schimbarea de pliere (pe axa x) și valoarea p (pe axa y). fus este gena modificată cel mai semnificativ la nivelul ARNm (marcat cu roșu) (C) Cuantificarea proteomului prin spectrometrie de masă fără etichete. Cuantificarea fără etichete a fost efectuată așa cum s-a descris anterior. 43 Expresia mediană a proteinelor din creierul WT (axa y) este reprezentată grafic față de expresia din fus -/- creier (axa x) pe scara log. Proteinelor care nu au fost detectate într-una din probe li s-a atribuit o valoare arbitrară de 22. Fusul este printre proteinele cele mai exprimate care au fost detectate în WT și lipsesc din knockout (marcate cu roșu).
Fundalul genetic afectează utilizarea alternativă 3'UTR
În timpul analizei datelor de secvențiere a ARN-ului, am observat că unele gene prezintă variații mari în expresia genei în rândul fraților (replicate biologice), independent de genotipul fus (Fig. S3). Peștele zebră nu poate fi menținut ca linii consangvinizate din cauza depresiei consangvinizate, 26 și fundalul liniei noastre este mixt (AB × TU). Am argumentat că efectele genetice de fond pot prevala asupra efectelor subtile ale knockout-ului fus. Pentru a aborda această problemă mai sistematic, am ales să ne concentrăm asupra utilizării alternative a site-ului de poliadenilare, care este relativ simplu de cuantificat și vizualizat. Am folosit isoSCM 27 pentru a detecta recent izoformele 3'UTR și le-am cuantificat expresia diferențială. Apoi, am numit polimorfisme cu nucleotide unice (SNPs) din datele ARN-Seq, profitând de o acoperire suficientă a citirii peste 3'UTRs. Într-adevăr, izoformele 3′UTR diferențiale de sus au urmat adesea distribuția SNP-urilor, și nu genotipul fus (Fig. 3). Astfel, suntem capabili să detectăm expresia diferențială a izoformei în datele noastre RNA-Seq, dar utilizarea izoformei pare să fie mai puternic influențată de fondul genetic local decât de expresia Fus.
Exemple de utilizare alternativă 3'UTR în diferite medii genetice. Sunt afișate capturi de ecran din browserul genomului IGV. Genele relevante sunt slc37a4b, pompa și imp3. Pentru comoditate, piesele de acoperire RNA-Seq sunt afișate în albastru doar pentru firul minus. SNP-urile numite care au fost filtrate (vezi metodele) sunt reprezentate de bare colorate deasupra liniei genetice. Albastrul închis sunt variante homozigote, barele roșii închise sunt heterozigote; referințele homozigote sau apelurile absente nu sunt afișate. Pista isoSCM prezintă alternative putătoare 3'end-uri numite de isoSCM. Scala pentru acoperirea RNA-Seq este normalizată la 1.000.000/(număr total de citiri) și este aceeași pentru toate piesele (afișate în gri între paranteze drepte în dreapta).
Embrionilor knock-out fusion le lipsesc defecte motoneuronale
Morfologia și atingerea motoneuronului au evocat răspunsul de evadare al larvelor knockout fus. (A) Imagini confocale ale trunchiurilor larvelor de 36 CP exprimând DsRed specific neuronului. 28 Larvele de knockout Fus zigotice materne prezintă morfologia generală a axonului motoneuronului primar normal, care nu se distinge de WT. Se arată proiecția de intensitate maximă a unei stive confocale; bara de scară este de 100µm. (B) Răspunsul evadat prin atingere a larvelor 2dpf (48-52hpf). Distanțele de înot în mm pentru urmele individuale ale larvelor sunt reprezentate. Valoarea P este din testul Kruskal-Wallis. n indică numărul de larve testate pentru fiecare grup.
Discuţie
Am folosit pește zebră învechit (AB × TU) pentru a genera animale knockout fus. Această strategie a fost benefică, deoarece embrionii din fondul TU injectat cu Cas9 foarte concentrat și ARN-ghid au arătat o supraviețuire slabă. Cu toate acestea, am observat că fundalul genetic poate influența puternic expresia genelor și utilizarea alternativă a izoformelor. Aceste efecte ar fi putut asigura modificări ușoare ale expresiei rezultate din pierderea Fus. În plus, datele noastre sugerează că efectele injecțiilor cu morfolino se pot manifesta diferit în funcție de fondul genetic al embrionilor. În concordanță cu aceasta, mai multe modele de șoareci knockout FUS prezintă un spectru larg de fenotipuri, care variază de la letalitatea postnatală în linii mai consangvinizate până la adulți supraviețuitori cu defecte morfologice și comportamentale ușoare într-o linie exterioară. 18-20 Întrucât densitatea ridicată a SNP-urilor și natura extrădată a peștilor zebră sunt mai asemănătoare cu cea de la oameni decât cea din liniile de șoarece consangvinizate, peștele zebră poate reprezenta un model foarte util pentru a studia efectele Fus sau ale oricărei alte proteine, în contextul variației genetice naturale.
Datele brute RNAseq sunt depuse în GEO (> GSE85554). Datele Proteomics au fost depuse la ProteomeXchange Consortium prin depozitul partener PRIDE cu identificatorul setului de date PXD004876e.
Metode
Metode extinse pot fi găsite în materialul suplimentar.
Creșterea și tulpinile de pește
Peștele a fost întreținut, crescut și pus în scenă așa cum s-a descris anterior. 35 Dacă nu se specifică altfel, în toate experimentele a fost utilizat un amestec de tulpină AB și TU. Toate experimentele au fost efectuate în conformitate cu legea germană privind bunăstarea animalelor (licențe 23 177-07/G 13-5-0-087 (eliminări CRISPR-Cas9), 23177-07/A 15-5-5001 OES (cleme pentru aripioare). Knockout-urile CRISPR-Cas9 au fost generate așa cum s-a descris. 24
secvențierea ARNm
Bibliotecile de ARN poliadenilate specifice catenelor au fost preparate folosind trusa de pregătire a bibliotecii de ARN catenar TruSeq și secvențiate pe un Illumina HiSeq2000 utilizând protocolul de citire 2 × 100bp. Citirile de secvențiere a ARN au fost mapate la genomul peștilor zebră (Zv10) folosind alinierea STAR (versiunea 2.4.1d 36). Citirea numărului de tabele a fost produsă cu featureCounts (v. 1.4.6 37) utilizând adnotarea genei bazei de date Ensembl (versiunea 80). Genele exprimate diferențial au fost numite folosind pachetul R DESeq2. 38
Spectrometrie de masa
Pregătirea și măsurarea probei de spectrometrie de masă s-a făcut așa cum este descris 39, cu excepția unei coloane C18 de 50 cm și a fost utilizat un gradient LC de 240 minute. Eșantioanele au fost analizate cu MaxQuant (versiunea 1.5.2.8 40) față de baza de date Uniprot zebrafish furnizată (39.559 intrări) cu setări standard, cu excepția valorilor LFQ bazate pe peptide unice. Pentru analiza datelor, informațiile despre peptidele identificate au fost extrase din tabelul de dovezi. La nivelul proteinei, valorile medii ale LFQ au fost calculate pentru fiecare afecțiune (WT, fus -/-) din fișierul grupurilor de proteine și reprezentate grafic ca dispersie în R utilizând pachetul ggplot2. 41
Imagine confocală
Trunchiurile de pește zebră au fost realizate pe microscopul confocal Leica SP5. Imagini de proiecție maximă au fost generate cu software-ul Fiji. 42
Injecții cu morfolino
Fus morfolinul (GGCCATAATCATTTGACGCCATGTT) 21 și controlul morfolinului cu 5 nepotriviri (GCCCATAATGATTTCACGGCATCTT) au fost de la Genetools. Aproximativ 1 nl picătură de 1mM Fus morfolin a fost injectată în embrioni în stadiul cu 1 celulă.
- Articolul complet 167 Caracterizarea fizico-chimică a amilazelor L
- Interacțiunea genetică cu contraceptivele poate determina creșterea în greutate
- Mâncați un mic dejun mare pentru a slăbi Acest vechi adagiu nutrițional nu este; susținut de știința genetică
- Dezvoltarea și caracterizarea unui nou hidrogel pe bază de galactomanan și κ-caragenan -
- Calculator FFMI Calculați-vă potențialul muscular genetic