Institutul de cercetare pentru medicină fizico-chimică, Moscova, Rusia
Autorul corespunzator: LN Ikryannikova, Institutul de Cercetare pentru Medicină Fizico-Chimică al Ministerului Sănătății Publice al Federației Ruse, str. 119992 Malaya Pirogovskaya, 1a, Moscova, Rusia
Institutul de cercetare pentru medicină fizico-chimică, Moscova, Rusia
Institutul de cercetare pentru medicină fizico-chimică, Moscova, Rusia
Agenția Națională de Farmacologie Clinică și Farmacie, Moscova, Rusia
Agenția Națională de Farmacologie Clinică și Farmacie, Moscova, Rusia
Institutul de cercetare pentru medicină fizico-chimică, Moscova, Rusia
Agenția Națională de Farmacologie Clinică și Farmacie, Moscova, Rusia
Agenția Națională de Farmacologie Clinică și Farmacie, Moscova, Rusia
Institutul de cercetare pentru infecțiile copiilor, Saint Petersburg, Rusia
Universitatea Medicală de Stat din Sankt Petersburg, Sankt Petersburg, Rusia
Institutul de cercetare pentru medicină fizico-chimică, Moscova, Rusia
Institutul de cercetare pentru medicină fizico-chimică, Moscova, Rusia
Autorul corespunzator: LN Ikryannikova, Institutul de Cercetare pentru Medicină Fizico-Chimică al Ministerului Sănătății Publice al Federației Ruse, str. 119992 Malaya Pirogovskaya, 1a, Moscova, Rusia
Institutul de cercetare pentru medicină fizico-chimică, Moscova, Rusia
Institutul de cercetare pentru medicină fizico-chimică, Moscova, Rusia
Agenția Națională de Farmacologie Clinică și Farmacie, Moscova, Rusia
Agenția Națională de Farmacologie Clinică și Farmacie, Moscova, Rusia
Institutul de cercetare pentru medicină fizico-chimică, Moscova, Rusia
Agenția Națională de Farmacologie Clinică și Farmacie, Moscova, Rusia
Agenția Națională de Farmacologie Clinică și Farmacie, Moscova, Rusia
Institutul de cercetare pentru infecțiile copiilor, Saint Petersburg, Rusia
Universitatea Medicală de Stat din Sankt Petersburg, Sankt Petersburg, Rusia
Institutul de cercetare pentru medicină fizico-chimică, Moscova, Rusia
Abstract
Introducere
Streptococcus pneumoniae, Streptococcus mitis, Streptococcus pseudopneumoniae și Streptococcus oralis sunt specii strâns înrudite de v iridan g roup s treptococci (VGS) care colonizează cavitatea bucală umană; cu toate acestea, proprietățile lor patogene diferă semnificativ. In timp ce S. pneumoniae este un agent patogen uman major asociat cu pneumonie dobândită în comunitate, meningită și otită medie, alți reprezentanți ai acestui grup sunt comensali și pot provoca infecții numai atunci când au acces la fluxul sanguin sau la o gazdă imunocompromisă. Laboratoarele clinice trebuie să poată diferenția cu exactitate S. pneumoniae din alte VGS întâlnite frecvent în probele clinice pentru a facilita terapia antimicrobiană adecvată. Cu toate acestea, metodele convenționale fenotipice, cum ar fi morfologia coloniilor, solubilitatea biliară și testarea sensibilității la optochină, precum și sistemele comerciale (API 20 Strep și Vitek 2; bioMe′rieux, Marcy l'Etoile, Franța), nu oferă întotdeauna o identificare precisă 1- 4 .
O varietate de gene au fost utilizate ca ținte pentru o discriminare bazată pe PCR a VGS: pneumolizină (stratul) 5, autolizină (lytA) 6, antigen de suprafață pneumococic A (câineA) 7 și fragmentul ADN cu funcție necunoscută Spn9802 8. Cu toate acestea, aplicarea acestei strategii este complicată de rapoartele care Streptococ mitis și Streptococcus oralis adăpostește genele care codifică autolizina și pneumolizina 9-11. Analiza secvenței a genelor ARNr 16S, de asemenea, nu poate fi aplicată pentru discriminarea VGS datorită asemănării 99% în compoziția nucleotidică a genelor ARNr 16S la aceste bacterii 12, 13. Analiza secvenței de rnpB (Subunitatea ARN a endonuclează P), sifon (superoxid dismutază dependentă de mangan), tuf (factor de alungire Tu), groESL (proteinele de șoc termic) și rpoGenele B (b - subunitatea ARN polimerazei bacteriene) sunt mai promițătoare, dar datele sunt insuficiente pentru concluziile finale.
În prezent, identificarea cea mai fiabilă a VGS poate fi realizată prin utilizarea echivalenței multimetrice a analizei (MLSA) 14. Această abordare se bazează pe construirea unui copac filogenetic pe secvențele concatenate a șapte fragmente de gene de menaj și afișarea relației clonale dintre tulpinile necunoscute în studiu și tulpinile stocate în bazele de date publice. http://viridans.emlsa.net/ permite ca tulpinile streptococice să fie atribuite speciilor din VGS. Corect S. pneumoniae identificarea poate fi realizată, de asemenea, utilizând baza de date mlst.net, care a fost concepută în principal pentru tiparea intraspecie a pneumococilor 15. Din păcate, MLSA este relativ scump și consumă mult timp.
Recent a fost sugerată profilarea directă a bacteriilor prin ionizare prin desorbție cu laser matricială asistată - spectrometria de masă în timp (MALDI - TOF MS) a fost sugerată ca instrument pentru identificarea rapidă a diferitelor bacterii. Din păcate, folosind baza de date Biotyper 3.0 (Bruker Daltonics, Bremen, Germania) streptococ speciile mitis/oralis pot fi identificate eronat ca S. pneumoniae, datorită asemănării excepționale a spectrelor lor de masă 16-19 .
În acest studiu, s-au aplicat o serie de algoritmi de clasificare matematică pentru a sorta spectrele de masă ale VGS prin selectarea unui set de vârfuri de masă care discriminează izolatele caracterizate fenotipic și genetic ale diferitelor specii VGS. Din aceste clase, au fost generate o serie de modele de clasificare și comparate de parametrii de sensibilitate și specificitate. În cele din urmă, modelele de succes au fost testate orb pe cele selectate aleator S. pneumoniae și S. mitis tulpini.
Metode
Tulpini
Un total de 62 VGS au fost incluse în studiu. Treizeci și patru dintre ele au fost identificate în studiul nostru anterior ca fiind S. mitis iar trei ca. S. oralis 20. Douăzeci și cinci de izolate au fost identificate ca fiind S. pneumoniae pe baza solubilității biliare, a sensibilității la optochină și a rezultatelor pozitive ale testului ‘Slidex ® pneumo-kit’ (bioMerieux ®, Marcy - l’Etoile, Franța). Pentru S. pneumoniae izolate, serotipurile au fost determinate folosind antiseruri obținute de la Staten Serum Institute (Copenhaga, Danemarca) conform recomandărilor producătorului. Toate izolatele VGS au fost depozitate la -80 ° C în flacoane CRYOBANK ™ (Copan, Italia).
Înainte de analiza spectrometriei de masă și studiile genetice, izolatele au fost subculturate pe agar Columbia (Oxoid Ltd, Basingstoke, Marea Britanie) cu adăugare de 5% sânge de oaie (incubație peste noapte la 35 ° C în aer cu 5% CO2) și teste fenotipice (solubilitate biliară, sensibilitatea la optochină și testul „Slidex® pneumo-kit”) au fost repetate. Un test de sensibilitate la optochină a fost efectuat folosind discurile standard de diagnosticare cu optochină (Centrul de Cercetare în Farmacoterapie, St Petersburg, Rusia), în aer cu 5% CO2.
Analiza genelor: scheme MLST și MLSA
Pentru manipulările genetice, ADN-ul streptococic a fost extras folosind kitul „DNA - express” („Lytech” Ltd, Moscova, Rusia), în conformitate cu instrucțiunile producătorului. MLST și MLSA au fost efectuate așa cum au fost descrise de Enright și Spratt 15 și de Bishop și colab. 14, respectiv. Rezultatele au fost analizate folosind bazele de date MLST (http://www.mlst.net) și MLSA (http://viridans.emlsa.net/).
Achiziționarea datelor MALDI TOF
Pentru analiza spectrometriei de masă MALDI - TOF, două până la trei colonii izolate de culturi bacteriene proaspete (18 ore) au fost preluate cu o buclă de plastic sterilă de 1,0 μl (FL Medical, Torreglia, Italia) și suspendate în 300 μL de apă pură (Fluka, St. Louis, MO, SUA). După precipitarea cu etanol (900 pl), peleta a fost tratată cu un amestec de acid formic/acetonitril. Extractele bacteriene (1 μl) au fost reperate pe un eșantion țintă MALDI, suprapuse cu 1 μL din matrice (soluție saturată de acid alfa-ciano-4-hidroxi cinamic (CHCA) în 50% acetonitril/2,5% TFA) și uscate în aerul. Spectrele de masă au fost înregistrate pe un spectrometru de masă Microflex MALDI-TOF (Bruker Daltonics) echipat cu un laser N2 337 nm. Nu mai puțin de patru spectre de masă au fost obținute din fiecare izolat, cu un total de 250 de fotografii pe spectru (50 de fotografii în fiecare din cele cinci poziții diferite la fața locului). Pentru inspecția spectrelor vizuale a fost utilizat software-ul FlexAnalysis 2.4 (Bruker Daltonics). Software-ul BioTyper 3.0 (Bruker Daltonics) a fost utilizat pentru compararea și identificarea spectrelor.
Prelucrarea datelor cu software-ul ClinProTools
Software-ul ClinProTools 2.1 (Bruker Daltonics) a fost utilizat pentru recunoașterea modelelor peptidice. Acest software a fost furnizat inițial ca parte a soluției de sistem ClinProt ™ pentru căutarea biomarkerilor în fluidele corpului uman (ser, plasmă, urină, salivă, lichid spinal cerebral etc.) asociate cu diferite boli. Permite măsurarea și vizualizarea diferențelor de peptide și proteine în spectrele de masă ale diferitelor probe 21. Operațiile de pretratare a spectrelor, de culegere a vârfurilor și de calcul ale vârfurilor au fost efectuate folosind modul standard. Modelele de clasificare au fost generate folosind algoritmii algoritmului genetic (GA), rețelei neuronale supravegheate (SNN) și algoritmilor QuickClassifier (QC).
Pentru fiecare model, capacitatea de recunoaștere (RC), numărul relativ de spectre clasificate corect pentru modelul dat sub constrângerea că toate datele testate sunt utilizate anterior pentru determinarea modelului și validarea încrucișată (CV), o măsură cantitativă pentru au fost calculate fiabilitatea unui model care poate fi folosit pentru a prezice cum se va comporta un model în viitor.
Validarea externă a fost efectuată pe izolatele selectate orbește; s-au determinat valorile sensibilității și specificității.
Rezultate
Analiza MLST și MLSA
MLST a fost efectuat pe 25 de izolate de S. pneumoniae, 34 din S. mitis și trei din S. oralis. Toate S. pneumoniae izolatele erau susceptibile la optochină; nouă dintre ele aparțin serotipului 23F, cinci la 6B, cinci la 19F, două la 18, iar serotipurile 14, 19A, 9L și 35F au fost prezentate de câte un izolat fiecare.
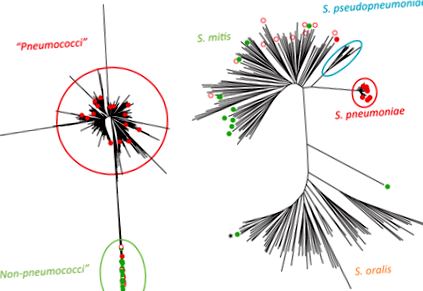
Dendrograma care descrie relații clonale între izolate este prezentată în Fig. 1 (a). Pentru doi S. oralis izolează, nu a reușit să amplifice dormi locus genetic, astfel încât aceste tulpini au fost aruncate.
Majoritatea izolatelor din grupul „pneumococ” (24 din 25) au căzut în clusterul pneumococic (Fig. 1a, în partea de sus a imaginii), în timp ce izolatele mitis și oralis au format clusterul separat („non-pneumococic”). Pentru izolatele din grupul „non-pneumococic”, s-a constatat că alelele tuturor fragmentelor genetice sunt cel puțin unul sau mai multe procente diferite de oricare cunoscută pentru alela pneumococului. Doar o tulpină (43 741) din grupul „pneumococi” a căzut în mod neașteptat în grupul „non-pneumococ”. Această tulpină a fost în mod stabil susceptibilă la optochină și a fost serotipată ca 19F. Apoi am construit arborele filogenetic folosind ClonalFrame v. 1.1 algoritmi, care ne permit să luăm în considerare posibilele recombinații la populațiile bacteriene; distribuția tulpinilor părea să fie aceeași (datele nu sunt prezentate).
Izolate selectate aleatoriu din pneumococ (n = 13) și de la non-pneumococice (n = 22) clustere, inclusiv două S. oralis izolează negativ pe dormi locus, au fost analizate suplimentar prin schema MLSA.
O dendrogramă bazată pe secvențe concatenate de șapte gene de menaj din izolate incluse în studiu și 244 de tulpini ale grupului mitis (S. mitis, S. pneumoniae, S. pseudopneumoniae și S. oralis) stocate în baza de date http://viridans.emlsa.net/ a fost creată (Fig. 1b). Toate izolatele din grupul „pneumococic” așa cum sunt definite prin analiza MLST au căzut în S. pneumoniae analiza MLSA, în timp ce 21 din 22 de izolate din grupul „non-pneumococic”, inclusiv izolatul 43 741, au căzut în S. mitis grup. A fost un izolat din grupul „non-pneumococic”, care s-a grupat la S. oralis ramură, împreună cu două S. oralis izolate pentru care dormi locusul nu a reușit să se amplifice în schema MLST (acestea sunt marcate de asteriscuri în figura 1b).
O parte din izolatele identificate de MLSA ca fiind S. pneumoniae (n = 13) sau S. mit (n = 21) au format principalele grupuri pentru generarea de modele de clasificare. Trei tulpini atribuite S. oralis a format grupul ‘oralis’. Alte izolate incluse în studiu (11 S. pneumoniae și 14 S. mitis) au format grupurile auxiliare pentru validarea modelelor.
Achiziționarea datelor de spectrometrie de masă și generarea modelului de clasificare
Aproximativ 120 de vârfuri cu rapoarte semnal/zgomot mai mari de 5 au fost detectate între m/z 2000 și 20 000 în spectrele de masă ale 62 de izolate studiate.
Toate izolatele clusterului „pneumococic” (vezi Fig. 1) au fost identificate de către software-ul BioTyper ca fiind S. pneumoniae cu un scor spectral de ≥2,3. Majoritatea izolatelor grupului „non-pneumococic” au fost identificate ca fiind S. pneumoniae cu un scor de ≥2,0, iar unele izolate au fost identificate ca S. pneumoniae sau S. oralis, sau S. pseudopneumoniae, sau S. cristatus, cu scoruri nu mai mici de 2.0.
Trei izolate ale S. oralis cluster (Fig. 2) au fost, de asemenea, atribuite S. pneumoniae/S. oralis/S. pseudopneumoniae/S. cristatus specii.
Mai mult, spectrele de masă obținute din tulpinile grupului principal, 13 din S. pneumoniae și 21 din S. mitis, au fost comparate utilizând software-ul ClinProTools 2.1. Un număr de modele diferite de clasificare au fost generate utilizând algoritmii GA, SNN și QC. RC, CV, sensibilitatea și specificitatea modelelor sunt prezentate în Tabelul 1.
| GA | 100,00 | 100,00 | 100,0 | 98,6 |
| SNN | 100,00 | 98,65 | 100,0 | 100,0 |
| QC | 100,00 | 99,46 | 100,0 | 100,0 |
| QC - 3a A Un model generat de trei vârfuri (6949, 9876 și 9975 m/z) conferind greutatea semnificativă clasificării. | 98,40 | 98,44 | 97,9 | 100,0 |
- A Un model generat de trei vârfuri (6949, 9876 și 9975 m/z) conferind greutatea semnificativă clasificării.
În modelul GA, numărul de vârfuri poate fi definit de utilizator. Variantând acest parametru, am creat un model în care 17 vârfuri au dat 100% valori RC și CV (Tabelul 1). Numărul de vârfuri din ambele modele, SNN și QC, a fost detectat automat. Șase vârfuri au fost determinate în modelul SNN și cinci în modelul QC. O valoare CV de 100% a fost atinsă numai pentru modelul GA, în timp ce în alte modele a fost puțin mai mică. Rețineți că validarea încrucișată este o variantă a validării automate în timpul generării modelului: o mică parte din toate spectrele sunt lăsate în afara modelului și a analizei cluster, apoi aceste spectre sunt clasificate și se determină numărul de predicții de clasă corecte și greșite. Această procedură se repetă de mai multe ori, iar predicțiile de clasă corecte și greșite sunt acumulate pentru fiecare clasă.
Au existat trei vârfuri (6949, 9876 și 9975 m/z) care conferă greutățile statistice maxime, care au fost găsite în fiecare model generat. FIG. 2 (a) prezintă un exemplu de vârf de m/z 6949, care contribuie semnificativ la modelul de discriminare a speciilor: se poate observa că acest vârf minor apare în aproape toate S. mitis izolează spectrele de masă și este absent în S. pneumoniae spectre de masă. Distribuția bidimensională pentru cele două vârfuri (6949 și 9975 m/z) este prezentată în Fig. 2 litera (b). În conformitate cu această diagramă, chiar și aceste două vârfuri au fost suficiente pentru discriminarea convingătoare a speciilor streptococice. De fapt, forțarea numai a acestor trei vârfuri (6949, 9876 și 9975 m/z) într-un model (desemnat ca QC - 3 în Tabelul 1) a fost urmată de o scădere minoră a valorilor RC și CV (98,40% și 98,44%, respectiv).).
Fiecare dintre modele a fost evaluat orbește folosind 25 de izolate din grupul auxiliar (11 de pneumococi și 14 de S. mitis). Toate cele trei modele au prezentat valori de sensibilitate și specificitate de aproape 100% (Tabelul 1).
Numarul S. oralis izolate (n = 3) a fost insuficient pentru a crea un model de încredere. Cu toate acestea, calcule scurte au arătat că toți clasificatorii au putut discrimina S. pneumoniae din S. oralis izolate, dar nu S. mitis din S. oralis izolate.
Discuţie
Specii precise - identificarea la nivel a izolatelor VGS este importantă atât din punct de vedere al practicii, cât și pentru înțelegerea mecanismelor patogene ale speciei respective. Pentru streptococii grupului mitis, identificarea speciilor este deosebit de dificilă. Numeroase abordări care utilizează diferitele trăsături ale speciilor fenotipice nu oferă o sensibilitate bună și nu pot fi considerate fiabile.
Introducerea profilării proteinelor bacteriene bazate pe MALDI - TOF a îmbunătățit semnificativ procesul de identificare a microorganismelor în practica de rutină. Această abordare se bazează pe investigarea spectrelor complexe de masă ale peptidelor și proteinelor mici, care conțin unice m/z „Semnături” pentru diferite microorganisme datorită variațiilor inerente ale maselor lor și au permis identificarea și discriminarea cu succes a unui spectru larg de organisme. Primele încercări de a discrimina VGS folosind MALDI - TOF MS promiteau 16, 19, 22, dar mai târziu a devenit clar că Biotyper 3.0 (Bruker) nu este în măsură să rezolve problema 17, 18, 23, 24. Foarte recent, a fost introdus un nou sistem Vitek MS (bioMérieux) și a prezentat rezultate relativ bune 25 .
- Noul serviciu Celesio din Marea Britanie permite pacienților să primească tratamente de salvare
- Clinici gratuite și bazate pe venit Seaside CA.
- A reușit cineva să scape de mama lor Netmums
- DD Fitness Produse de slăbit și influențatori online care îi mint pe adepții lor; Donegal Daily
- Cuplul fructitar crede că dieta lor îi ajută să rămână tineri Metro News