Karen S. Sarkisyan
1 Institutul de Chimie Bioorganică Shemyakin-Ovchinnikov, Miklukho-Maklaya 16/10, 117997 Moscova, Rusia
2 Academia de Stat Nizhny Novgorod, Minin Sq. 10/1, 603005 Nijni Novgorod, Rusia
3 Institutul Tehnologic Central European, Universitatea Masaryk, Brno, Republica Cehă
4 Programul de Bioinformatică și Genomică, Centrul de Reglare Genomică (CRG), Institutul de Știință și Tehnologie din Barcelona, 88 Dr. Aiguader, 08003 Barcelona, Spania
5 Universitat Pompeu Fabra (UPF), 08003 Barcelona, Spania
Dmitry A. Bolotin
1 Institutul de Chimie Bioorganică Shemyakin-Ovchinnikov, Miklukho-Maklaya 16/10, 117997 Moscova, Rusia
3 Institutul Tehnologic Central European, Universitatea Masaryk, Brno, Republica Cehă
Margarita V. Meer
4 Programul de Bioinformatică și Genomică, Centrul de Reglare Genomică (CRG), Institutul de Știință și Tehnologie din Barcelona, 88 Dr. Aiguader, 08003 Barcelona, Spania
5 Universitat Pompeu Fabra (UPF), 08003 Barcelona, Spania
Dinara R. Usmanova
4 Programul de Bioinformatică și Genomică, Centrul de Reglare Genomică (CRG), Institutul de Știință și Tehnologie din Barcelona, 88 Dr. Aiguader, 08003 Barcelona, Spania
5 Universitat Pompeu Fabra (UPF), 08003 Barcelona, Spania
6 Institutul de Fizică și Tehnologie din Moscova, Institutskiy pereulok 9, Dolgoprudny, 141700, Rusia
Alexander S. Mishin
1 Institutul de Chimie Bioorganică Shemyakin-Ovchinnikov, Miklukho-Maklaya 16/10, 117997 Moscova, Rusia
2 Academia de Stat Nizhny Novgorod, Minin Sq. 10/1, 603005 Nijni Novgorod, Rusia
George V. Sharonov
1 Institutul de Chimie Bioorganică Shemyakin-Ovchinnikov, Miklukho-Maklaya 16/10, 117997 Moscova, Rusia
7 Facultatea de Medicină, Universitatea de Stat din Moscova, av. Lomonosov. 31/5 119192 Moscova, Rusia
Dmitry N. Ivankov
4 Programul de Bioinformatică și Genomică, Centrul de Reglare Genomică (CRG), Institutul de Știință și Tehnologie din Barcelona, 88 Dr. Aiguader, 08003 Barcelona, Spania
5 Universitat Pompeu Fabra (UPF), 08003 Barcelona, Spania
8 Laboratorul de Fizică a Proteinelor, Institutul de Cercetare a Proteinelor din Academia Rusă de Științe, str. Institutskaya 4, Pushchino, regiunea Moscovei, 142290, Rusia
Nina G. Bozhanova
1 Institutul de Chimie Bioorganică Shemyakin-Ovchinnikov, Miklukho-Maklaya 16/10, 117997 Moscova, Rusia
Mihail S. Baranov
1 Institutul de Chimie Bioorganică Shemyakin-Ovchinnikov, Miklukho-Maklaya 16/10, 117997 Moscova, Rusia
9 Pirogov Russian National Research Medical University, Ostrovitianov 1, Moscova, 117997, Rusia
Onuralp Soylemez
4 Programul de Bioinformatică și Genomică, Centrul de Reglare Genomică (CRG), Institutul de Știință și Tehnologie din Barcelona, 88 Dr. Aiguader, 08003 Barcelona, Spania
5 Universitat Pompeu Fabra (UPF), 08003 Barcelona, Spania
Natalya S. Bogatyreva
4 Programul de Bioinformatică și Genomică, Centrul de Reglare Genomică (CRG), Institutul de Știință și Tehnologie din Barcelona, 88 Dr. Aiguader, 08003 Barcelona, Spania
5 Universitat Pompeu Fabra (UPF), 08003 Barcelona, Spania
8 Laboratorul de Fizică a Proteinelor, Institutul de Cercetare a Proteinelor din Academia Rusă de Științe, str. Institutskaya 4, Pushchino, regiunea Moscovei, 142290, Rusia
Peter K. Vlasov
4 Programul de Bioinformatică și Genomică, Centrul de Reglare Genomică (CRG), Institutul de Știință și Tehnologie din Barcelona, 88 Dr. Aiguader, 08003 Barcelona, Spania
5 Universitat Pompeu Fabra (UPF), 08003 Barcelona, Spania
Evgeny S. Egorov
1 Institutul de Chimie Bioorganică Shemyakin-Ovchinnikov, Miklukho-Maklaya 16/10, 117997 Moscova, Rusia
Maria D. Logacheva
9 Pirogov Russian National Research Medical University, Ostrovitianov 1, Moscova, 117997, Rusia
10 A.A. Institutul Kharkevich pentru probleme de transmitere a informațiilor, Academia Rusă de Științe, Moscova, Rusia
11 Departamentul de Bioinformatică și Bioinginerie, Universitatea de Stat din Moscova, Moscova, Rusia
Alexey S. Kondrashov
11 Departamentul de Bioinformatică și Bioinginerie, Universitatea de Stat din Moscova, Moscova, Rusia
12 Departamentul de Ecologie și Biologie Evolutivă, Universitatea din Michigan, Ann Arbor, MI, SUA
Dmitry M. Chudakov
1 Institutul de Chimie Bioorganică Shemyakin-Ovchinnikov, Miklukho-Maklaya 16/10, 117997 Moscova, Rusia
3 Institutul Tehnologic Central European, Universitatea Masaryk, Brno, Republica Cehă
Ekaterina V. Putintseva
1 Institutul de Chimie Bioorganică Shemyakin-Ovchinnikov, Miklukho-Maklaya 16/10, 117997 Moscova, Rusia
3 Institutul Tehnologic Central European, Universitatea Masaryk, Brno, Republica Cehă
Ilgar Z. Mamedov
1 Institutul de Chimie Bioorganică Shemyakin-Ovchinnikov, Miklukho-Maklaya 16/10, 117997 Moscova, Rusia
3 Institutul Tehnologic Central European, Universitatea Masaryk, Brno, Republica Cehă
Dan S. Tawfik
13 Departamentul de chimie biologică, Institutul de Științe Weizmann, Rehovot 76100, Israel
Konstantin A. Lukyanov
1 Institutul de Chimie Bioorganică Shemyakin-Ovchinnikov, Miklukho-Maklaya 16/10, 117997 Moscova, Rusia
2 Academia de Stat Nizhny Novgorod, Minin Sq. 10/1, 603005 Nijni Novgorod, Rusia
Fyodor A. Kondrashov
4 Programul de Bioinformatică și Genomică, Centrul de Reglare Genomică (CRG), Institutul de Știință și Tehnologie din Barcelona, 88 Dr. Aiguader, 08003 Barcelona, Spania
5 Universitat Pompeu Fabra (UPF), 08003 Barcelona, Spania
14 Institution Catalana de Recerca i Estudis Avançats (ICREA), 23 Pg. Lluís Companys, 08010 Barcelona, Spania
Date asociate
Abstract
Am analizat peisajul de fitness local al avGFP prin estimarea nivelurilor de fluorescență ale genotipurilor obținute prin mutageneză aleatorie a secvenței avGFP (Figura 1). Am folosit sortarea celulelor activate de fluorescență și secvențiat întreaga regiune de codificare GFP pentru a testa fluorescența a mii de genotipuri create prin mutageneză aleatorie a secvenței de tip sălbatic (Informații suplimentare S2 și date extinse Fig. 1). Am aplicat mai multe strategii pentru a minimiza eroarea estimării noastre de fluorescență (informații suplimentare S3.4 și S4.4), care a fost estimată din mii de măsurători independente ale secvenței de tip sălbatic (rata de eroare fals negativă 0,08%) și genotipuri care încorporează mutații cunoscute pentru a elimina fluorescența (rata de eroare fals pozitivă 0,24%). Setul nostru de date final a inclus 56.086 secvențe de nucleotide unice care codifică 51.715 secvențe de proteine diferite. Procedura noastră a introdus în medie 3,7 mutații per secvență de gene, iar majoritatea genotipurilor testate conțineau mai multe, până la 15, mutații cu sens. Totuși, deoarece numărul total de secvențe posibile crește exponențial odată cu numărul de mutații, fracțiunea de secvențe eșantionate a fost mică pentru secvențele care conțin mai mult de două mutații (Tabelul 1 de date extinse). Am folosit aceste date pentru a analiza peisajul de fitness local al GFP analizând impactul mutațiilor simple, duble și multiple.
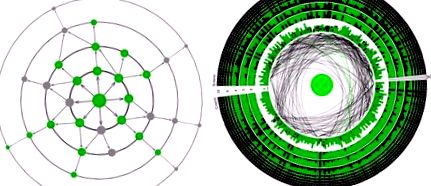
A, AvGFP de tip sălbatic (centru) și majoritatea mutanților unici (cercul cel mai interior) fluoresc în verde. Genotipurile cu mutații multiple pot prezenta epistaze negative, cu combinații de mutații neutre care creează fenotipuri non-fluorescente (gri) sau epistoză pozitivă, prin care o mutație într-un genotip non-fluorescent restabilește fluorescența. b, Secvența GFP aranjată într-un cerc, fiecare coloană reprezentând un situs de aminoacizi. În primul cerc, intensitatea culorii pătratelor indică luminozitatea unei singure mutații la locul corespunzător în raport cu tipul sălbatic, arătat în centru. Site-urile cu interacțiuni epistatice pozitive și negative între perechi de mutații sunt conectate prin linii verzi, respectiv negre. În cercuri mai îndepărtate de centru, reprezentând genotipuri cu mutații multiple, fracția coloanei colorată verde (negru) reprezentând fracția de genotipuri corespunzătoare fluorescenței mari (scăzute) dintre toate genotipurile testate cu o mutație la acel loc. Foarfecele indică locul restricției.
Distribuția efectelor de fitness ale mutațiilor individuale missense a fost testată prin compararea distribuției fluorescenței secvențelor de aminoacizi avGFP de tip sălbatic, marcate prin diferite coduri de bare moleculare și a distribuției fluorescenței secvențelor care au o singură mutație (Informații suplimentare S4.1). Am constatat că cel puțin 75% dintre mutații au avut un efect dăunător asupra fluorescenței, inclusiv 9,4% din mutațiile unice conferind o scădere> de 5 ori a fluorescenței, dar pentru multe mutații efectul a fost mic (Figura 2a). În consecință, genotipurile cu mutații multiple de sens au fost mai susceptibile de a avea fluorescență scăzută și majoritatea genotipurilor care poartă cinci sau mai multe mutații de sens au fost non-fluorescente (date extinse Fig. 2). Mutațiile cu un efect puternic asupra fluorescenței au locuit, de preferință, în siturile care au codificat reziduurile de aminoacizi orientate intern spre cromofor (Figura 2b, c), care este în concordanță cu datele privind alte proteine cu privire la preferința mutațiilor dăunătoare pentru a viza reziduurile îngropate 9, 11 - 13. Impactul mutațiilor asupra fluorescenței a fost corelat pozitiv cu conservarea sitului (date extinse Fig. 3a, coeficientul de corelație a rangului Spearman 0,40 ± 1,44 × 10 −10) și mai puțin probabil să se regăsească în secvențe ortologice (date extinse Fig. 3b). Încă,
10% din stările mutante care conferă un fenotip non-fluorescent au fost totuși fixate în evoluția pe termen lung (date extinse Fig. 3b), indicând faptul că epistoză afectează peisajul de fitness avGFP 16 .
A, Distribuțiile fluorescenței pentru 2442 secvențe de tip sălbatic măsurate independent (gri), pentru 1114 mutații simple (albastru) și fracția estimată de mutații neutre (alb). b, Mutațiile cu un singur sens care influențează puternic fluorescența (violeta) au avut tendința să apară în locuri cu reziduuri orientate intern., c, afișat pe un fir beta selectat al structurii GFP.
A, O reprezentare ipotetică a epistazei negative și pozitive în funcție de numărul de mutații unice din avGFP. b,Fracția genotipurilor non-fluorescente observate (roșu) și fracția preconizată a genotipurilor non-fluorescente calculate ca suma impactului log asupra fluorescenței mutațiilor individuale (albastru). c, Distribuțiile epistazei pentru epistazele negative și pozitive de forță diferită, cu rata de descoperire falsă așteptată prezentată în gri.
Într-un peisaj unidimensional, fitnessul este o funcție monotonă a unei variabile intermediare, numită potențialul de fitness 21, 22, care este suma impacturilor mutațiilor individuale. Am folosit regresia multiplă având în vedere o funcție de fitness neepistatică prin care log-fluorescența, F, este egală cu predictorul liniar, potențialul de fitness, p, astfel încât F = f (p) = p. Acest model simplu, non-epistatic, a explicat doar 70% din varianța eșantionului inițial (σ 2 = 1,12 și σ 2 = 0,34 înainte și după aplicarea modelului, respectiv). Folosind varianța celor 2442 măsurători de fluorescență de tip sălbatic am estimat că
1% din varianța eșantionului inițial poate fi atribuită zgomotului (σ 2 = 0,0097), indicând faptul că restul de 29% din varianța eșantionului nu poate fi explicat fără epistază.
Cea mai simplă formă a unei funcții de fitness epistatică este atunci când fitness-ul este o funcție monotonă neliniară de p 21, 22. Lipsa genotipurilor cu fluorescență intermediară (date extinse Fig. 5a) sugerează că peisajul de fitness avGFP poate fi descris printr-o funcție de fitness asemănătoare trunchierii 23. Prin urmare, am modelat F ca o funcție sigmoidă a lui p, ceea ce a explicat 85% din varianța eșantionului inițial (σ 2 = 0,17). O funcție de fitness mai complexă în formă de sigmoidă rafinată cu o abordare de rețea neuronală (Informații suplimentare S4.6) a explicat 93,5% din varianța eșantionului inițial (σ 2 = 0,065, date extinse Fig. 5), confirmând că peisajul de fitness poate fi în mare parte reprezentat de o funcție de prag unidimensională (Figura 4), care poate apărea din contribuția comună a mutațiilor la stabilitatea proteinelor 8, 13, 14, 20, 24. Fluorescența medie a mutanților unici ai avGFP în funcție de destabilizarea proteinei prezisă, ΔΔG, relevă un prag în jur de 7-9 kcal/mol (Figura 4). Interesant este faptul că valoarea ascunsă găsită de rețeaua neuronală artificială pentru mutanți singuri s-a corelat cu ΔΔG prezis (Figura 4, Date extinse Fig. 5f), confirmând o influență probabilă a stabilității proteinelor asupra naturii epistazei în avGFP. Funcția de fitness prag face o treabă remarcabil de bună în aproximarea întregului peisaj de fitness explicând
95% din toate varianțele. Cu toate acestea, atunci când luăm în considerare rata de eroare a setului nostru de date, estimăm că cel puțin 0,3% din genotipuri nu pot fi explicate prin funcția de fitness prag (Informații suplimentare S4.5 și date extinse Fig. 5d) reprezentând cazuri de epistază multidimensională 2, 5, 7 .
Fluorescența mediană a GFP cu mutații unice în funcție de efectul lor asupra energiei de pliere prevăzute (∆∆G), cu SD, suprapusă cu funcția de fitness asemănătoare sigmoidului obținută independent, prevăzută de rețeaua neuronală (linia portocalie).
Rata normalizată de evoluție convergentă către stări de aminoacizi ancestrali terminali și reconstituiți pentru fiecare coș de distanță (puncte gri). Probabilitatea așteptată (linia portocalie) și observată în datele experimentale (puncte portocalii) ca o singură mutație să rămână fluorescentă pe măsură ce secvența acumulează alte substituții. Probabilitatea așteptată (linia verde) și observată (punctele verzi) ca o mutație non-fluorescentă să devină fluorescentă cu divergența secvenței. Barele reprezintă un interval de încredere în proporție binomială (nivel de încredere 68%).
Congruența largă a datelor noastre cu prevalența epistazei din evoluția pe termen lung sugerează că forma peisajului local de fitness poate fi extrapolată la o scară mai mare. Cu toate acestea, epistaza între siturile care codifică reziduurile cu o interacțiune directă în structura proteinelor a fost rară, contrastând cu observarea unor astfel de cazuri în evoluția pe termen lung 16 și cu o analiză a mutației domeniului RRM 12. Astfel, peisajul de fitness local care acoperă câteva mutații dintr-un singur vârf de fitness poate fi aproximat printr-un prag unidimensional de funcție de potențial de fitness, cu toate acestea, această funcție de fitness simplă poate să nu fie adecvată pentru a descrie peisaje de fitness care încorporează creste de fitness care conectează secvențe de ortologi mai divergenți. 27. Natura peisajelor globale de fitness, în special interacțiunea dintre scările locale și globale, rămâne de explorat.
- Peisaj local de fitness al proteinei fluorescente verzi Natura
- Masa musculară - Se poate învinui shake-ul proteic pentru că nu pierde în greutate - Schimb fizic de stivă
- Gustări cu conținut ridicat de proteine pentru pierderea în greutate - Fitness St Pete l St Pete Fitness și Pierdere în greutate - Fitness
- Herbsgreen Japan Red Reishi Ciupercă feliată (1 kg) - Nutriție verde nouă
- Jillian Michaels Fitness Apk Plătit Deblocat ultima descărcare Android