Abstract
fundal
Pakistanul acoperă o zonă geografică cheie din istoria omenirii, fiind atât parte a regiunii râului Indus, care a acționat ca una dintre leagănele civilizației, cât și ca o legătură între Eurasia de Vest și Asia de Est. Această regiune este locuită de o serie de grupuri etnice distincte, cel mai mare fiind punjabi, Pathan (Pakhtuns), sindhi și Baloch.
Rezultate
Am analizat primul genom etnic masculin Pathan prin secvențierea acestuia la o acoperire de 29,7 ori folosind platforma Illumina HiSeq2000. Un total de 3,8 milioane de variații ale nucleotidelor unice (SNV) și 0,5 milioane de indeli mici au fost identificate prin compararea cu genomul uman de referință. Dintre SNV-uri, 129.441 au fost noi și 10.315 SNV-uri sinonime au fost găsite în 5.344 de gene. SNV-urile au fost adnotate pentru consecințe asupra sănătății și boli cu risc ridicat, precum și pentru posibile influențe asupra eficacității medicamentului. Am confirmat că genomul Pathan prezentat aici este un reprezentant al acestui grup etnic, comparându-l cu un grup de asiatici centrali din panourile HGDP-CEPH tipizate pentru
650 k SNP-uri. ADNmt (H2) și haplogrupul Y (L1) ale acestui individ erau, de asemenea, tipice regiunii sale geografice de origine. În cele din urmă, reconstituim istoria demografică de către PSMC, care evidențiază o creștere recentă a dimensiunii efective a populației, compatibilă cu amestecul dintre descendenții europeni și asiatici așteptați în această regiune geografică.
Concluzii
Prezentăm o secvență de genom întreg și analize ale unui etan Patan din provincia nord-vestică a Pakistanului. Este o resursă utilă pentru a înțelege variația genetică și migrația umană pe întregul continent asiatic.
fundal
Tehnologia secvențierii se îmbunătățește rapid, cu o reducere drastică a costurilor sale [1]. Aceste progrese rapide ne-au extins foarte mult înțelegerea diversității genetice umane și a istoriei populației [2], permițându-ne să investigăm variante cu consecințe asupra sănătății și deschizând calea către medicina personalizată [3]. Studiile de asociere pe scară largă a genomului (GWAS) au caracterizat funcția a mii de SNV-uri comune, dar există încă milioane de variante rămase neexplorate [4]. Prin urmare, secvențierea întregului genom este necesară pentru un studiu detaliat al variantelor genomice rare. Un număr de consorții internaționale au început secvențierea întregului genom al unor panouri mari, inclusiv Proiectul 1000 Genomi (www.1000genomes.org), Proiectul Genomului Personal (www.personalgenomes.org) și 100 genomuri Malay [5]. Aceste consorții, precum și mai multe proiecte geografic mai restrânse, își propun să înțeleagă aspectele funcționale ale variantelor comune și unice la om. În viitor, ne putem aștepta ca toate grupurile etnice distincte să aibă secvențe genomice.
Pakistanul se află la o intersecție a sub-continentului indian în est, a statelor din Asia Centrală în vest și a Chinei spre nordul său. Are o istorie socio-religioasă-culturală unică, în plus față de o serie de grupuri etnice și lingvistice, cum ar fi punjabi, Pathan (Pakhtuns), sindhi și Baloch (Fișier suplimentar 1: Figura S1) [6]. În timp ce un număr dintre aceste grupuri au fost incluse în panourile genetice de tipare a microsateliților și SNP-urilor [7], doar un individ masculin pakistanez de origine etnică necunoscută a fost secvențiat până acum (Fișier suplimentar 1: Figura S2) [8]. Aici raportăm prima secvență a întregului genom și analiza unui bărbat Pathan (cetățean pakistanez). Variațiile genomice incluzând variații de nucleotide unice (SNV), inserții mici și deleții (indels) și regiuni de variație a numărului de copii (CNVR) au fost identificate prin alinierea secvenței genomului Pathan la genomul uman de referință (hg19). Variantele au fost apoi adnotate și scanate pentru funcții asociate împreună cu SNV-uri care ar putea modula răspunsul la medicament. S-au investigat posibile SNV-uri non-sinonime dăunătoare (nsSNV) pentru potențialul efect asupra farmacocineticii și farmacodinamicii medicamentelor. În plus, au fost utilizate mai multe abordări analitice pentru a evalua influența contribuțiilor ancestrale în genomul Pathan (PTN).
rezultate si discutii
Secvențierea genomului și identificarea variantelor
ADN-ul extras din sânge a fost secvențiat cu citiri de capăt pereche de 90 bp folosind secvențierul Illumina HiSeq2000, producând 1.069.127.687 citiri. Un total de 83,3 Gb de secvențe au fost generate și aliniate la genomul uman de referință (fără Ns, 2.861.343.702 bp), acoperind 98,2% din genomul de referință la o adâncime medie de 28,5 × (Fișier suplimentar 2: Tabel S1).
Am identificat un total de 3.813.440 SNV-uri, dintre care 3.683.999 (96,6%) au fost raportate în baza de date dbSNP [9] și 129.441 au fost noi (Tabelul 1), care au fost comparate în continuare cu numărul de variante noi ale altor genomi individuali din literatura 1: Figura S3) [10-19]. Au existat 1.272.912 SNV homozigoti și 2.540.528 heterozigoți. Un total de 18.547 SNV-uri au fost găsite în regiunile de codificare a secvenței ADN (CDS), 25.481 în regiunile netraduse de 3 ’(UTR) și 4.969 în UTR de 5’. Un total de 10.315 SNV-uri în 5.344 gene au fost non-sinonime (nsSNV-uri).
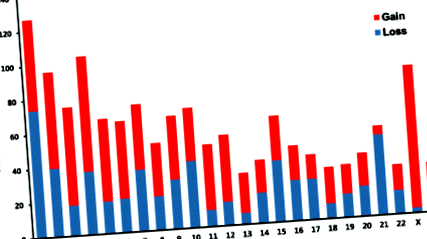
Au fost observate un total de 504.276 indeli scurți (până la ± 20 baze), dintre care 306.128 au fost găsite în regiunile intergenice, 237 în regiunile CDS și 193.308 în regiunile intron. În plus, s-au găsit 1.503 CNVR, dintre care 713 au fost clasificate ca duplicate și 790 ca șterse, afectând 2.364 gene suprapuse (Fișier suplimentar 3: Tabelul S2). Un total de 65 CNVR nu fuseseră descrise anterior în baza de date a variantelor genomice (DGV; http://projects.tcag.ca/variation/). Figura 1 arată numărul de CNVR câștigate și pierdute în fiecare cromozom. ANNOVAR a fost utilizat pentru analiza detaliată a adnotării CNVR-urilor pentru a identifica genele asociate cu aceste regiuni (Fișier suplimentar 4: Tabel S3).
Copiați regiunile de variație a numărului din genomul Pathan. Copierea numărului de variații ale numărului distribuite în fiecare cromozom.
Clasificarea funcțională și relevanța clinică a variantelor
Toate cele 10.315 nsSNV găsite în genomul Pathan au fost examinate în continuare pentru posibilele lor efecte funcționale folosind metode de predicție de calcul (SIFT și Polifen2), rezultând 43 nsSNV în 43 de gene clasificate ca fiind dăunătoare funcțional (Fișier suplimentar 5: Tabelul S4). În plus, nsSNV-urile au fost adnotate folosind ClinVar pentru relevanța lor clinică și am constatat că 31 de SNV-uri codificatoare sunt asociate cu mai multe boli (Fișier suplimentar 6: Tabel S5). De remarcat în special sunt un SNV (rs1049296, Pro570Ser) în TF gena [20], care afectează susceptibilitatea Alzheimer; Ser217Leu in ELAC2 genă (rs4792311), care este implicată în susceptibilitatea genetică la cancerul de prostată ereditar [21]. Rata cancerului de prostată este scăzută în Pakistan (3,8%) [22], comparativ cu americanii și caucazianul [23]. Trei SNV-uri de codare activate GHRLOS (rs696217, Leu72Met), SERPINE1 (rs6092, Ala15Thr) și PPARG (rs1801282, Pro12Ala) care au toate legături cu obezitatea [24-26]. Aproximativ 22,2% dintre pakistanezi sunt raportați ca fiind obezi, ceea ce este aproape de european (
24%) și populațiile Statelor Unite
De asemenea, am găsit trei SNV patogeni în gene asociate cu părul, pielea și pigmentarea: EDAR (rs3827760, Val370Ala), SLC45A2 (rs16891982, Phe374Leu) și TYR (rs1042602, Ser192Tyr) [30-32]. În plus, am detectat un SNV (rs17822931, Gly180Arg) în ABCC11, care este responsabil pentru ceară umedă care a fost găsită și în genomul pakistanez PK1 [33].
Una dintre variantele (rs1065852, Pro34Ser) din CYP2D6 gena este responsabilă de metabolismul deficitar al debrisoquinei, un medicament blocant adrenergic utilizat pentru tratamentul hipertensiunii [34]. De asemenea, două SNV-uri în TPMT Se știe că (rs1142345, Tyr240Cys și rs1800460, Ala154Thr) au un efect patogen și duc la deficiența tiopurinei metiltransferazei (TPMT) [35,36]. Mai mult, două nsSNV (rs2056899 și rs140980900) de CYP4A22 și GGT5 s-au găsit gene din calea metabolismului acidului arahidonic (fișa suplimentară 7: Tabelul S6). Acidul arahidonic din corpul uman provine de obicei din surse animale dietetice, cum ar fi carne, ouă și lactate. Carnea este o parte importantă a dietei Pathan, de obicei consumată cel puțin o dată pe zi, adesea sub formă de kabab (carne tocată prăjită în ulei) sau curry [37].
Analiza genomică comparativă a fost făcută folosind genomul Pathan (PTN) și celălalt genom publicat anterior pakistanez (PK1). Variantele non-sinonime din genomul Pakistanului (PK1) au fost adnotate pentru investigarea bolilor asociate. Din
8.000 nsSNV-uri au fost găsite doar 37 de variante (trei noi) legate de anumite tulburări. Opt SNV relevante clinic au fost detectate suprapuse cu genomul Pathan (PTN). Nu am găsit variante deteriorate responsabile de Alzheimer, obezitate și boli legate de inimă, așa cum am găsit în genomul Pathan (PTN). Un SNV (rs1057910; CYP2C9) a fost observat în genomul PK1, care este cunoscut pentru răspunsul Wafarin. Mai mult, o mutație patogenă (rs1169305) a fost observată în HNF1A genă care poate deveni o cauză a diabetului la individul PK1.
Majoritatea variantelor relevante clinic adoptate în acest studiu au fost descrise inițial la populațiile caucaziene. În timp ce acest rezultat ar putea fi o consecință a afinităților genomice ale genomului Pathan cu alte populații caucaziene, ar putea reflecta, de asemenea, o prejudecată din cauza majorității activității GWAS efectuate asupra populațiilor caucaziene [38]. Prin urmare, pentru autentificare va fi necesar un studiu de cohortă în populația pakistaneză.
Analiza farmacogenomică
Comparație cu alte persoane Patan
Am investigat cât de reprezentativ era genomul nostru Pathan pentru acel grup etnic, comparându-l cu alți douăzeci și doi de indivizi Pathan din grupul HGDP-CEPH [7], care fusese tipat pentru
650 k SNV-uri, împreună cu încă 190 de persoane din alte opt populații din Asia de Sud (pakistaneză) din același grup. Analiza amestecului a fost efectuată pe baza a 643.281 SNV (subțiat pentru a evita LD). Am considerat apartenența la cluster de la STRUCTURĂ (de la K = 2 la K = 5), compoziția genomului Pathan (PTN) s-a încadrat în variabilitatea observată în cadrul eșantionului Pathan din HGDP (Figura 2). În mod similar, într-un grafic de scalare multi-dimensională (MDS), genomul Pathan a intrat în ceilalți indivizi Pathan (Fișier suplimentar 1: Figura S4). Luate împreună, aceste două rezultate confirmă faptul că genomul Pathan prezentat în această lucrare este reprezentativ pentru grupul etnic Pathan. Aceste rezultate sunt, de asemenea, în conformitate cu strămoșii auto-raportați ai subiectului, toți bunicii săi venind din Afganistan în Khyber Pakhtunkhwa (Pakistan).
Rezultatele amestecului individului Pathan (PTN) la alte grupuri etnice din Asia de Sud. Rezultatele amestecului pentru K = 2 și K = 5 pentru individul Pathan combinat cu opt genomi etnici din setul de date HGDP. Analiza s-a bazat pe 643.281 de SNV-uri. Fiecare individ este reprezentat de o linie verticală, împărțită în segmente colorate care reprezintă coeficienți de apartenență la subgrupuri.
Analize ADNm și cromozom Y.
Genomul mitocondrial complet al individului Pathan a fost generat prin maparea citirilor acestuia la secvența de referință Cambridge (rCRS) revizuită [45]. Conținutul de adenină și timină (AT) al genomului a fost de 55,5%, în timp ce conținutul de guanină și citozină (GC) a fost de 44,5%. Un total de 57 SNV au fost găsite în genomul mitocondrial Pathan, dintre care 13 nu fuseseră raportate anterior. Variantele au fost apoi mapate cu MitoVariome [46] pentru a identifica haplogrupul mitocondrial al individului nostru Pathan. Un total de 14 SNV au fost diagnosticate pentru haplogrupul H2, despre care sa susținut că este de origine exclusiv caucaziană, iar apariția sa marginală în Pathans reflectă amestecul [47].
Conținutul AT și GC al cromozomului Y au fost 39,87%, respectiv 60,13%. Au fost identificate 13.724 SNV-uri, dintre care 4.423 au fost noi. SNV-urile cromozomiale Y observate au fost adnotate ca markeri pentru haplotipul L1 al cladei L. Haplogrupul L are o frecvență ridicată în Pakistan (14%) în comparație cu India (6,3%), Turcia
4%) și caucazieni
Analiza istoriei demografice
Am dedus istoria demografică a Patanului utilizând modelul pereche secvențial coalescent Markovian (PSMC) [51] (Figura 3) și l-am comparat cu un grup de populații la nivel mondial bazat pe un număr de genomi HGDP [52]. După cum sa raportat anterior, toate populațiile au o istorie demografică similară între 1 milion și 200 km în urmă. De acum 200km până acum 20kyr, Patanii urmează o traiectorie similară cu alte populații asiatice și europene, cu o dimensiune efectivă a populației dedusă mai mică decât populațiile africane, reflectând blocajul din Africa. În ultimii 20 de ani, Pathan arată o explozie a dimensiunii efective a populației, contemporană cu alte populații eurasiatice, dar cu o amplitudine mult mai mare. Mărimea efectivă foarte mare a populației reflectă probabil amestecul dintre liniile europene și asiatice, dând naștere la Patani moderni (așa cum sugerează și analiza ADNmt și a cromozomului Y), mai degrabă decât o creștere efectivă a mărimilor recensământului.
Dimensiuni istorice inferioare ale populației prin analiza coalescentă marchoviană secvențială în perechi. Analiza PSMC (Pairwise Sequentially Markovian Coalescent) a fost efectuată pentru a reconstitui istoria demografică a populației din Pathans, comparativ cu un set de 11 genomi HGDP din întreaga lume (Africa: Dinka, Yoruba, Mandenka, Mbuti, San; Asia: Dai, Han; Europa: franceză, sardă; Oceania: papuană; America: Karitiana).
Concluzii
Aici vă prezentăm, pentru prima dată, întregul genom al unui individ Pathan dintr-o provincie de nord-vest (Khyber Pakhtunkhwa) din Pakistan. Analiza noastră oferă o vedere detaliată a diversității genomului Pathan și clasificarea funcțională a variantelor și impactul acesteia în farmacogenomică. Este necesară o analiză pe scară largă a diverselor genomi pentru a ajuta cercetătorii din întreaga lume să înțeleagă diversitatea genetică și clasificarea funcțională a variantelor, împreună cu trăsăturile farmacogenomice și medicamentele asociate care ar fi utilizate ca medicină personalizată.
Metode
Selectarea subiectului și declarația etică
Acest studiu a fost realizat în conformitate cu Declarația de la Helsinki și a fost aprobat de Fundația de Cercetare a Genomului (GRF) de către Institutional Review Board cu IRB-REC-2011-10-003. Consimțămintele semnate au fost obținute de la participantul la acest studiu și consimțământul membrilor familiei sale cu privire la publicarea întregului conținut al informațiilor despre genom și fenotip, precum și informații personale de identificare (precum vârsta, sexul și locația).
Există cazuri documentate ale membrilor familiei sale cu hipertensiune, probleme cardiace, tulburări neuro, diabet și obezitate. Tatăl său a fost diagnosticat cu tulburări cardiovasculare, hipertensiune și Alzheimer. Mama sa are osteoartrita, iar bunicii au murit din cauza infarctului, a cancerului și a hipertensiunii.
Surse de date
Genomul de referință UCSC (hg19, februarie 2009), versiunea dbSNP 137 și adnotările genomului, au fost descărcate din baza de date (www.genome.ucsc.edu). Genomii din grupul HGDP-CEPH de 190 de persoane aparțin a opt populații din Asia de Sud (Balochi, Brahui, Burusho, Hazara, Kalash, Makrani, Pathan și Sindhi), care fuseseră tipizate pentru
650 k SNV-uri au fost preluate din baza de date disponibilă publicului.
Extracția ADN-ului
ADN-ul genomic a fost extras din limfocitele sanguine arteriale ale unui bărbat pakistanez Pathan, în vârstă de treizeci de ani, care locuiește în provincia de nord-vest a Pakistanului. QIAamp DNA Blood Mini Kit a fost utilizat pentru extragerea ADN-ului din sânge (Qiagen). Nanodrop Infinite F200 de la Tecan a fost utilizat pentru a evalua puritatea ADN-ului, 1,7% electroforeză pe gel de agaroză pentru a confirma dimensiunea ADN-ului (prezența ADN-ului cu greutate moleculară mare) și fluorometrul Invitrogen Qubit pentru a determina concentrația ADN-ului.
Analiza citogenetică
Cariotiparea a fost efectuată cu limfocite de sânge periferic cultivate folosind tehnici standard, iar banda GTG a fost utilizată pentru a identifica aberațiile cromozomiale, care este utilă pentru identificarea bolilor genetice prin reprezentarea fotografică a întregului complement cromozomial [53]. Nu s-au găsit anomalii cromozomiale evidente în analiza citogenetică prin imagistica cromozomială a cariotipului în bandă G (Fișă suplimentară 1: Figura S5).
Pregătirea bibliotecii și secvențierea întregului genom
Două biblioteci cu capăt pereche au fost preparate din 1,1 μg de ADNc folosind trusa de pregătire ADN Illumina TruSeq, urmând protocolul standard al Illumina (Paired-end Library Preparation Kit, Illumina, SanDiego, CA, SUA). Tunderea ADNc a fost făcută folosind seria Covaris S (Covaris, MS, SUA). După repararea finală, coada A și ligarea adaptorului, ADN-ul în intervalul 500-600 bp a fost purificat dintr-un gel de agaroză 2%. ADN-ul a fost apoi îmbogățit cu PCR pentru un total de zece cicluri. Mărimea corectă a ADN-ului a fost apoi confirmată cu Aganalent Bioanalyzer, urmată de cuantificarea qPCR cu reactivi Roche Light Cycler 480 II și reactivi Kapa Biosystems.
Generarea cluster a fost efectuată pe un Illumina cBot și bibliotecile au fost secvențiate pe un Illumina HiSeq 2000 urmând protocolul Paired-End. Secvențele pot fi accesate la NCBI SRA, cu numărul de acces SRA092047. Restul analizei noastre a fost inițiat din fișierele FASTQ furnizate de suita de software CASAVA din analiza în aval a Illumina.
Cartarea și alinierea la referința genomului
Secvențele genomului au fost aliniate cu genomul uman de referință (hg19) folosind Burrows-Wheeler Aligner (BWA; versiunea 0.5.9) [54] și SAMtools 0.1.16 [55] cu opțiunile implicite, cu excepția „aln -t 3 -l Opțiuni 45 -k 2 ”. Fișierele de aliniere au fost apoi îmbinate într-un singur fișier BAM, marcat pentru duplicate folosind Picard 1.59 (http://picard.sourceforge.net) și scorurile de calitate de bază au fost recalibrate folosind Genome Analysis Toolkit (GATK v1.4) [56].
SNV-uri, indels scurte și apeluri CNV-uri
SNV-urile și indel-urile mici variind de la 1 la 20 de baze au fost identificate folosind Genome Analysis Toolkit (GATK v1.4) cu HARD_TO_VALIDATE: MQ0 ≥ 4 și (MQ0/(1.0 × DP))> 0.1), 2) QualFilter = QUAL 2 mai mare decât 0.1 cu orice alt SNV din aceeași fereastră). Componentele MDS au fost obținute folosind opțiunea PLINK mds-plot bazată pe matricea de distanță identitate-după-stat (IBS). Analiza amestecului a fost efectuată folosind programul STRUCTURE pentru a identifica prezența unei relații ancestrale diverse a genomului Pathan (PTN) cu altele [63]. Am explorat valorile K de la 2 la 5 și am ales valoarea K care a dat cea mai mică eroare de validare încrucișată.
Analiză coalescentă secvențial markoviană
Am efectuat o analiză PSMC (Pairwise Sequentially Markovian Coalescent) pentru a reconstitui istoricul demografic al populației din Pathans [51]. Am comparat genomul Pathan cu un set de 11 genomi HGDP din întreaga lume (publicat de Meyer și colab.) [52]. Am folosit mai întâi samtools pentru a extrage genomii diploizi din fișierele lor BAM aliniate la hg19 și am exclus cromozomii sexuali și genomii mitocondriali deoarece sunt haploizi. În PSMC, am folosit opțiunile din linia de comandă -N25 -t15 -r5 -p "4 + 25 * 2 + 4 + 6" care au fost folosite cu succes în analize similare anterioare ale oamenilor și ale maimuțelor mari [64].
- Urine Trouble - Whole Dog Journal
- Unicode feliat subțire introduce noi emojiuri alimentare, Whole Foods ridică taxele furnizorilor și multe altele;
- Al 9-lea blog The Whole Life Challenge s-a încheiat
- Cele 7 obiceiuri sănătoase ale întregii vieți - Fairmont Miramar
- Ce este masa de degustare a alimentelor etnice