Ya jing Zhao
1 Școală de Medicină și Științe ale Vieții, Universitatea din Jinan, Academia de Științe Medicale Shandong, Jinan, China
2 Departamentul Laboratorului Clinic, Spitalul și Institutul pentru Cancer din Shandong, Prima Universitate Medicală din Shandong și Academia de Științe Medicale din Shandong, Jinan, China
Cântec Xingguo
3 Laboratorul cheie provincial de oncologie radiațională Shandong, spitalul și institutul de cancer Shandong, prima universitate medicală Shandong și Academia de științe medicale Shandong, Jinan, China
Limin Niu
1 Școală de Medicină și Științe ale Vieții, Universitatea din Jinan, Academia de Științe Medicale Shandong, Jinan, China
2 Departamentul Laboratorului Clinic, Spitalul și Institutul pentru Cancer din Shandong, Prima Universitate Medicală din Shandong și Academia de Științe Medicale din Shandong, Jinan, China
Youyong Tang
1 Școală de Medicină și Științe ale Vieții, Universitatea din Jinan, Academia de Științe Medicale Shandong, Jinan, China
2 Departamentul Laboratorului Clinic, Spitalul și Institutul pentru Cancer din Shandong, Prima Universitate Medicală din Shandong și Academia de Științe Medicale din Shandong, Jinan, China
Cântec Xianrang
2 Departamentul Laboratorului Clinic, Spitalul și Institutul pentru Cancer din Shandong, Prima Universitate de Medicină din Shandong și Academia de Științe Medicale din Shandong, Jinan, China
Li Xie
2 Departamentul Laboratorului Clinic, Spitalul și Institutul pentru Cancer din Shandong, Prima Universitate de Medicină din Shandong și Academia de Științe Medicale din Shandong, Jinan, China
Date asociate
Seturile de date analizate în acest manuscris nu sunt disponibile publicului. Solicitările de accesare a seturilor de date ar trebui să fie direcționate către moc.621@1002eix_l.
Abstract
Fundal: MiARN-urile exosomale circulante sunt potențiali biomarkeri neinvazivi pentru cancerul colorectal. Prezentul studiu și-a propus să valideze biomarkerii miRNA exosomali sensibili și specifici pentru diagnosticarea cancerului colorectal (CRC).
Pacienți și metode: Exosomii izolați din serul pacienților cu CRC și donatori sănătoși prin ultracentrifugare au fost caracterizați utilizând TEM, qNano și imunoblotare. Exosomii de la 2 donatori sănătoși și 4 pacienți cu CRC au fost supuși izolării ARN și secvențierea miARN. MiARN-urile exprimate diferit de la 165 de pacienți CRC primari și 153 de donatori sănătoși au fost confirmate de RT-qPCR.
Rezultate: Analiza datelor secvenței ARN a arătat că au fost selectate 29 miARN exosomale (20 reglate în jos și 9 reglate în sus) cu diferență> de 1,5 ori între pacienții cu CRC și donatorii sănătoși. Nivelurile serice de miR-99b-5p și miR-150-5p au fost în mod semnificativ subregulate la pacienții cu CRC în comparație cu donatorii sănătoși (p Cuvinte cheie: exosomi, cancer colorectal, miR-99b-5p, miR-150-5p, diagnostic
Introducere
Cancerul colorectal (CRC) este al treilea cel mai frecvent cancer la nivel mondial, cu 1,36 milioane de cazuri noi anual și aproximativ 700 de mii de decese (1, 2). Aproximativ, 50% dintre pacienții cu CRC sunt decedați ca urmare a depistării tardive a bolii avansate cu metastaze localizate sau la distanță (3). Aceste fenomene evidențiază și subliniază necesitatea identificării și dezvoltării biomarkerilor de screening robusti și ieftini, care sunt neinvazivi și facilitează detectarea timpurie a CRC, cum ar fi biopsia fluidă pe baza conținutului de sânge, inclusiv ADN fără celule (4), celule tumorale circulante (5) și exosomi (6).
Exosomii sunt cunoscuți ca vezicule extracelulare, cu diametrul de 50-150 nm, eliberate din diferite tipuri de celule (7-9), care sunt considerate mediatori critici ai comunicațiilor intercelulare, inclusiv livrarea de semnale biologice și încărcătura selectivă între diferite celule, prin urmare reglarea mai multor proceduri biologice (10, 11). Exosomii exercită, de asemenea, roluri pleiotrope în progresia cancerului, metastaze, modulații imune și rezistența la medicamente (12-16). Descoperirea critică a comunicării mediate de exosomi este transferul de informații genetice prin exosomi, cum ar fi ARN-uri mesager (ARNm) și microARN-uri scurte necodificatoare (miARN), către celule vecine sau organe îndepărtate. De exemplu, miARN-urile din exosomii de cancer sunt hormoni, care sunt vitale în medierea progresiei cancerului și a metastazelor, apărând astfel ca biomarkeri promițători pentru cancer (17).
În acest studiu, am utilizat secvențierea ARN-ului mic și PCR cantitativă pentru a căuta miARN diferențial exosomal între pacienții cu CRC și, respectiv, donatorii sănătoși. În consecință, au fost selectate miR-150-5p și miR-99b-5p exosomale și a fost analizată corelația lor cu caracteristicile clinice și eficiența diagnosticului pentru diagnosticul CRC; astfel, acestea au servit ca biomarkeri noi pentru CRC.
Materiale și metode
Pacienți
Un total de 169 pacienți cu CRC, 155 de donatori sănătoși și 20 de pacienți cu boli benigne au fost internați la Spitalul de Cancer din Shandong din septembrie 2017 până în iulie 2018. Exozomii de la 2 donatori sănătoși și 4 pacienți cu CRC au fost supuși secvențierii miRNA și 165 de pacienți cu CRC, exosomii de la alți 155 de donatori sănătoși și 20 de pacienți cu boală benignă au fost supuși verificării qPCR. Consimțământul informat scris a fost obținut de la toți subiecții. Stadializarea tumorală a fost estimată în conformitate cu AJCC Cancer Staging Handbook al American Joint Committee on Cancer, 2010. Toți pacienții nu au primit tratament antitumoral înainte de colectarea serului sau au suferit de alte boli endocrine, imune sau metabolice. Serurile au fost colectate de la 20/165 pacienți cu CRC, care au fost supuși unei intervenții chirurgicale după 2 luni. Caracteristicile pacienților și istoricul diabetului la pacienții cu CRC sunt prezentate în Tabelul 3.
Izolarea exozomilor
Exosomii au fost izolați folosind ultracentrifugare așa cum s-a descris anterior (25). Pe scurt, serul a fost centrifugat la 10.000 × g timp de 30 de minute la 4 ° C pentru a îndepărta resturile celulare, urmat de ultracentrifugare (Beckman Coulter, Brea, CA, SUA) la 100.000 × g timp de 2 ore la 4 ° C pentru precipitații exosomice. Apoi, sedimentul exosom a fost analizat prin microscopie electronică de transmisie (TEM), qNano și imunoblotare, secvențierea miARN și PCR în timp real.
Analiza TEM
TEM a fost efectuat pentru a identifica exosomii purificați. Peletele de exosom au fost transferate pe rețele într-o picătură de 50 μL de glutaraldehidă 1% timp de 5 minute și transferate într-o picătură de 100 μL de apă distilată și au lăsat grilele să stea timp de 2 minute. Apoi, grilele au fost plasate direct la o picătură de 50-l de soluție de uranil-oxalat (pH 7), timp de 5 minute și acoperite cu o farfurie de sticlă acoperită cu parafilm acoperită cu gheață anonică. Ulterior, grilele au fost spălate de șapte ori cu apă distilată timp de 2 minute fiecare și examinate folosind un microscop electronic cu transmisie JEM-1200EX (JEOL, Japonia) funcționat la 100 kV.
Senzor de impuls rezistiv reglabil (TRPS)
Mărimea nanoparticulelor a fost măsurată utilizând TRPS și pe qNano (Izon Science Ltd, Christchurch, Noua Zeelandă). Datele au fost analizate folosind Izon Control Suite v.3.3.2.2000 (Izon Science Ltd.).
Imunoblotarea
O cantitate echivalentă de proteine exosomale sau celulare a fost rezolvată prin SDS-PAGE și transferată în membranele PVDF (Millipore, Billerica, MA, SUA). Membranele au fost blocate cu lapte 5% în soluție salină tamponată Tris conținând 0,1% Tween 20 (TBST) timp de 1 oră și sondate peste noapte la 4 ° C cu anticorpi primari de iepure împotriva CD63, TSG101 și GM130, urmată de incubare cu HRP- anticorpi secundari conjugați (Proteintech) timp de 1 oră la temperatura camerei. Benzile imunoreactive au fost vizualizate utilizând reactivi de detectare ECL blotting (Bio-Rad, SUA) și au fost dezvoltate și fixate pe filme cu raze X.
Expresie diferențială miARN de seturi de date de secvență de ARN și analiză
Un total de 3 μg ARN din fiecare probă a fost utilizat ca material de intrare pentru generarea unei biblioteci mici de ARN. După generarea clusterului, bibliotecile au fost secvențiate pe o platformă Illumina HiSeq 2500/2000 (Illumina, SUA) și au fost generate citiri cu un singur capăt de 50 bp. După secvențiere, datele au fost supuse următoarelor analize și proceduri preliminare, care au fost efectuate de către Novogene Corporation: analiza controlului calității, citirea cartografierii la genomul homo spaiens, asamblarea transcriptomului, analiza potențialului de codificare, analiza conservatoare, predicția genei țintă, gena cuantificarea nivelului de expresie, analiza expresiei diferențiale și analiza de îmbogățire a Enciclopediei de gene și genomi (KEGG) de la Kyoto. Analiza expresiei diferențiale a două condiții/grupuri a fost efectuată utilizând pachetul DESeq R (1.8.3). Valorile P au fost ajustate folosind metoda Benjamini & Hochberg. Valoarea P corectată de 0,05 a fost setată ca prag pentru expresia diferențiată în mod implicit. Am folosit software-ul KOBAS pentru a testa îmbogățirea statistică a genei țintă candidate în căile KEGG. Distribuția hiper-geometrică non-centrală Wallenius bazată pe GOseq, care s-ar putea ajusta pentru distorsiunea lungimii genei, a fost implementată pentru analiza de îmbogățire a GO. Prezicerea genei țintă a miARN a fost efectuată RNAhybrid: (https://bibiserv.cebitec.uni-bielefeld.de/rnahybrid/); PITA: (genie.weizmann.ac.il/pubs/mir07/mir07_dyn_data.html); miRanda: (http://www.microrna.org/microrna/getMirnaForm.do) (Tabelul S1).
Izolarea ARN și PCR în timp real
ARN-urile totale au fost extrase cu reactiv TRIzol (Thermo Fisher Scientific, Carlsbad, CA, SUA) conform instrucțiunilor producătorului. ARN-ul extras a fost transcris invers în ADN complementar (ADNc) folosind trusa Mix-X miRNA First-Strand Synthesis Kit (TaKaRa Bio, Nojihigashi, Kusatsu, Japonia) conform instrucțiunilor producătorului. PCR în timp real a fost efectuat folosind TB-Green Premix Ex Taq II Reagent (TaKaRa Bio) conform instrucțiunilor producătorului. U6 a fost utilizat ca control intern (26). Fiecare probă a fost analizată în duplicat. Reacția PCR a fost evaluată prin analiza curbei de topire. Cuantificarea relativă a expresiei miARN a fost evaluată folosind metoda ΔCT (Ct miARN -Ct U6) așa cum a fost descris anterior (27).
Analize statistice
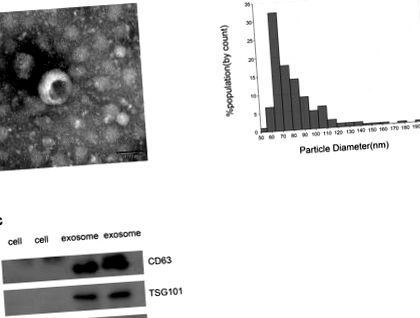
Analiza statistică a fost efectuată folosind SPSS 22.0 (IBM, Ehningen, Germania) și GraphPad Prism 6.0 (San Diego, CA, SUA). Comparațiile au fost efectuate folosind Mann-Whitney U sau testul t, iar diferența dintre valorile asociate a fost evaluată folosind un test t asociat. Comparații multiple au fost efectuate utilizând analiza unică a varianței (ANOVA) sau Kruskal - Wallis ANOVA unidirecțională. Curbele caracteristice de funcționare a receptorului (ROC) cu statisticile C corespunzătoare (zona de sub curbă, AUC), pe baza modelelor logistice, au fost utilizate pentru a determina punctele de tăiere corespunzătoare cu diagnosticul patologic tratat ca „etalonul de aur”. p Figura 1A) ilustrează morfologia tipică rotundă tip exosom cu diametrul de 50-150 nm de către TEM, care a fost în acord cu rezultatul de la qNano (Figura 1B). În plus, CD63 și TSG101, doi markeri de proteine bine cunoscuți (28, 29), sunt îmbogățiți în exozomi de la pacienții cu CRC, dar sunt nedetectabili în celulă (Figura 1C). Pe de altă parte, GM130 este un factor de legare asociat cu gigantina din compartimentul cis-Golgi utilizat în mod obișnuit ca control negativ pentru exosom (Figura 1C) (30). A fost detectat numai în celulă, dar nu și în exozomul CRC. Prin urmare, aceste rezultate au confirmat că veziculele izolate prin ultracentrifugare erau exosomi.
Identificarea exosomilor izolați. (A) Imaginea TEM a arătat date reprezentative ale exosomilor de la pacienții cu CRC cu diametrul tumorilor de 50-150 nm (bară de scală: 50 nm; tensiune înaltă (HV) = 100 kV). (B) Distribuția exosomilor cu diametrul de 50-150 nm; probele au fost obținute de la pacienții cu CRC pe baza sistemului qNano. (C) Analiza Western blot a CD63, TSG101 și GM130 ca markeri exosomali.
Profilul miRNA exosomal al pacienților cu CRC
Exosomii de la 2 donatori sănătoși și 4 pacienți cu CRC au fost supuși izolării ARN și secvențierea miARN. Un total de 1.145 miARN diferențiali au fost selectați între donatorii sănătoși și pacienții cu CRC prin analiza datelor de profilare a expresiei miARN brute (Figura 2A). Mai mult, 29 miARN (20 reglate în jos și 9 reglate în sus, Tabelele 1, 2) 2) au fost selectate pe baza diferenței de> 1,5 ori între cele două grupuri (Figura 2B). Analiza KEGG a demonstrat că genele țintă ale miARN-urilor exprimate diferit au fost atribuite în primul rând la 20 de căi, printre care căile de semnalizare a proteinei kinazei activate mitogen (MAPK) și căile de semnalizare a guanozinei monofosfat-protein kinază G (cGMP-PKG) au fost implicate în principal în miARN exosomal. funcții în CRC (Figura 2C).
Profilul mios ARN exosomal al pacienților cu CRC. (A) O hartă de căldură a fost generată după o analiză ierarhică supravegheată a clusterelor. Expresia diferențială a miARN este prezentată în roșu (reglare ascendentă) vs. albastru (reglare descendentă) (p Figura 3A). În plus, am validat stabilitatea miARN-urilor exosomale. Expresia miARN în exosomi a fost încă neschimbată la tratamentul cu RNază A (Figura 3B). Pe scurt, rezultatele arată că miR-99b-5p și miR-150-5p există în principal în exosomi, care protejează miRNA de tratamentul enzimatic. În testul de incubație la temperatura camerei, exosomii au fost menținuți la temperatura camerei timp de 0, 6, 12, 18, 24 h. Nu s-au găsit modificări semnificative pentru nivelurile de expresie ale miR-99b-5p și miR-150-5p la diferite puncte de timp (Figurile 3C, D).
- Modele dietetice și riscul apariției adenoamelor colorectale cancerul studiului sănătății femeilor negre
- Efectul kefirului asupra calității vieții pacienților tratați pentru cancer colorectal - PubMed
- Tiparele dietetice și cancerul colorectal rezultă dintr-un studiu canadian bazat pe populație Nutrition
- MicroARN-urile extracelulare circulante în sânge și salivă sunt o nouă sursă de biomarkeri în
- Semnături microbiene globale pentru cancerul colorectal - ScienceDaily