Abstract
Introducere
Obezitatea este o problemă majoră în Statele Unite, cu peste două treimi din adulți și o treime din adolescenți clasificați ca supraponderali sau obezi Ogden și colab. (2012). În timp ce excesul de greutate este un factor de risc pentru diabetul de tip 2, majoritatea persoanelor obeze nu dezvoltă diabet de tip 2 (T2D) (Boyle și colab. 2010; Eckel și colab. 2011; Grup de scriere pentru căutarea diabetului în grupul de studiu pentru tineri și colab. 2007). Adiponectina poate ajuta la explicarea riscului crescut de T2D la unii indivizi obezi. Adiponectina are proprietăți antiinflamatorii și sensibilizante la insulină, iar nivelurile scăzute de adiponectină preced dezvoltarea dezvoltării rezistenței la insulină și a T2D (Hotta și colab. 2001; Lindsay și colab. 2002). Mecanic, adiponectina facilitează acțiunile insulinei în țesuturile periferice prin activarea AMP kinazei și a p38 MAPK (Combs și colab. 2001; Yamauchi și colab. 2002). Cu toate acestea, efectele adiponectinei asupra sensibilității la insulină sunt mai relevante din punct de vedere clinic la persoanele obeze. La indivizii slabi Martin și colab. (2005) și șoareci Maeda și colab. (2002), concentrațiile de adiponectină nu sunt asociate cu sensibilitatea la insulină; totuși, în contextul obezității, concentrațiile de adiponectină arată o asociere puternică cu nivelurile de insulină (Maeda și colab. 2002; Martin și colab. 2005).
Adiponectina este codificată de ADIPOQ (NCBI GeneID 9370) situat la 3q27. ADIPOQ variațiile nucleotidice unice (SNV) au fost asociate cu T2D (p -2), rezistența la insulină (p -2) și adiponectina serică (p-8) (Vasseur și colab. 2002; Filippi și colab. 2004; Hivert și colab. 2008; Mackevics și colab. 2006; Mousavinasab și colab. 2006). Adiponectina se multimerizează rezultând patru forme oligomerice circulante: trimere (LMW), hexameri (MMW), greutate moleculară mare (HMW) și complexe cu greutate moleculară foarte mare (VHMW) complexe Tsao și colab. (2002). Complexele HMW și VHMW sunt cele mai active biologic Pajvani și colab. (2004). Până în prezent, au fost descrise cinci SNV cauzale (R55H, G84R, G90S, R112C și R131H). Aceste SNV sunt asociate cu hipoadiponectinemie, afectarea multimerizării adiponectinei și T2D (Waki și colab. 2003; Takahashi și colab. 2000; Jungtrakoon și colab. 2011). Cu toate acestea, aceste SNV sunt rare și nu reușesc să explice pe deplin variabilitatea adiponectinei, sugerând că există SNV cauzale neidentificate.
Astfel, obiectivul nostru este să identificăm noi și cunoscute ADIPOQ modificări de codificare și le raportează la formele oligomerice de adiponectină. Prin secvențiere ADIPOQ la adolescenții cu niveluri extreme de adiponectină serică (ridicată sau scăzută), ne-am îmbogățit grupul de studiu pentru non-sinonime ADIPOQ SNV-uri. În acest articol, vă prezentăm ADIPOQ variațiile regiunii de codificare și efectul lor asupra oligomerizării adiponectinei. În timp ce impactul acestor SNV poate fi mascat la indivizii slabi, odată validate în studii mecaniciste, aceste mutații ar putea fi incluse într-o metrică utilizată de medici pentru a consilia copiii cu risc crescut pentru riscul crescut de rezistență la insulină înainte de a deveni supraponderali insensibil.
Rezultate
Au fost identificate patru SNV-uri sinonime: un salt de inițiere a metioninei (c.130 G> A prescurtat ca MIS), P32L, R55C și Y111H (Figura 1). Fiecare SNV a fost prezent ca heterozigot și a fost găsit la un singur individ. Trei dintre cele 4 SNV (MIS, P32L și R55C) au fost identificate în rândul celor 10 (30% rata de succes) cu cel mai mic procent de 1 adiponectină din cohorta noastră de adiponectină serică foarte scăzută. În Exome Project al NHLBI, doar 21 non-sinonime ADIPOQ modificări au fost identificate la 5138 persoane: o rată de succes de 0,4%. Selectarea indivizilor cu cele mai scăzute niveluri de adiponectină la secvență a condus la o îmbogățire semnificativă statistic pentru modificările nesinonime în comparație cu proiectul de secvențiere NHLBI (valoarea exactă a testului Fisher -5).
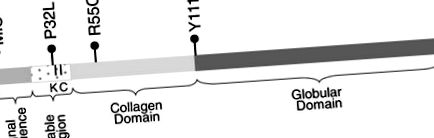
ADIPOQ structura genei și localizarea modificărilor de codificare identificate. MIS, P32L, R55C și Y111H. K este poziția unei lizine hidroxilate și glicozilate. C este poziția unei cisteine care formează legături disulfurice.
Persoanele cu SNV de codificare au prezentat eterogenitate fenotipică în ceea ce privește IMC și insulină (Tabelul 1), dar valorile glucozei au fost clinice normale și, prin urmare, nu au fost prezentate. Interesant este faptul că indivizii cu SNV adiponectinici și valori scăzute ale adiponectinei au afectat oligomerizarea, după cum reiese din Western blots (Figura 2) și ELISA oligomerice (Tabelul 2), chiar și atunci când contabilizează variabilitatea IMC și insulină.
Oligomerizarea adiponectinei în ser utilizând Western blotting pe geluri nereducătoare. C, control negativ al bufferului de încărcare numai. V, individ cu variație. M, control asortat. MM, control nepotrivit. Datorită concentrațiilor scăzute de adiponectină din unele probe, gelurile au fost administrate folosind două concentrații diferite (panoul A: 2 uL, panoul B: 4 uL).
Identificarea adiponectinei noi SNV: MIS
Participantul nostru cu MIS a avut cea mai mică adiponectină din cohortă; cu toate acestea, acest individ a prezentat adipozitate normală (CDC percentil BMI percentile Tabelul 3 In silico evaluarea rare ADIPOQ variații (SNV)
Identificarea adiponectinei SNV R55C
Participantul nostru cu R55C avea adiponectină scăzută și era rezistent la insulină și era obez (IMC ≥ CDC percentila 95). Acest SNV a fost raportat recent ca parte a Proiectului NHLBI Exome, dar nu au fost furnizate informații fenotipice. Western blot prezintă o lipsă de VHMW și puțină adiponectină HMW, cu o bandă LMW semnificativă la 2μL (Figura 2). La 4μL, se observă o bandă dublă LMW, indicând probabil un dimer, care nu a fost văzut în alte probe. Din ELISA, adiponectina HMW nu este detectabilă. În mod izbitor, scorurile SIFT/Polifen-2 pentru această mutație prezic un efect dăunător/probabil dăunător egal în mărime cu mutația cauzală R112C (Tabelul 3).
Identificarea adiponectinei SNV Y111H
Persoana cu acest SNV raportat anterior avea diabet de tip 1 și niveluri totale de adiponectină peste media cohortei noastre. Acest individ a fost slab și nu a prezentat diferențe în structurile oligomerice circulante în raport fie cu controalele ei de potrivire, fie cu cele de nepotrivire. Scorurile SIFT și PolyPhen-2 pentru această modificare prezic că Y111H este tolerat și benign (Tabelul 3).
Discuţie
Deoarece adiponectina este asociată cu riscul de T2D legat de obezitate și este puternic determinată de factori genetici, obiectivul nostru a fost să identificăm noi și cunoscute ADIPOQ modificări de codificare și relaționează relația lor cu formele oligomerice de adiponectină. Folosind o abordare extremă de fenotipare discordantă, patru heterozigoți non-sinonimi ADIPOQ au fost identificate modificări, dintre care două erau noi. Prin secvențierea indivizilor cu niveluri serice extrem de scăzute de adiponectină am îmbogățit proba pentru SNV-uri non-sinonime în comparație cu o probă secvențiată, fără a respecta nivelurile de adiponectină. Trei din cele patru SNV-uri au prezentat o oligomerizare afectată în concordanță cu efectul funcțional prezis bioinformatic. Odată validate biologic, aceste variații ar putea fi folosite pentru a identifica persoanele cu risc de rezistență la insulină și T2D legate de obezitate.
Y111H a fost raportat (rs17366743) (Vasseur și colab. 2002; Waki și colab. 2003; Kretowski și colab. 2005), cu o frecvență minoră a alelelor de 5,8%. În timp ce rapoartele anterioare sugerează că nivelurile de adiponectină au fost reduse cu acest SNV (Vasseur și colab. 2002; Kretowski și colab. 2005), individul cu această modificare a avut niveluri de adiponectină peste media pentru cohorta noastră, în concordanță cu nivelurile mai ridicate de adiponectină observate la indivizii cu tipul I diabet (Morales și colab. 2004; Frystyk și colab. 2005). Mai mult, acest SNV nu pare să modifice distribuția oligomerică a adiponectinei.
În această lucrare, raportăm identificarea a trei heterozigoți putativ funcționali ADIPOQ SNV după secvențierea a zece indivizi cu cea mai mică adiponectină din cohorta noastră. Având în vedere rata noastră mare de succes în identificarea SNV-urilor de codificare, secvențierea genelor candidate și o abordare extremă a fenotipului poate fi un mecanism excelent pentru a completa abordările actuale de secvențiere a exomului. Foarte important, abordarea noastră a identificat SNV-uri heterozigoți non-sinonimi rare; evaluarea acestor modificări heterozigote non-sinonime poate fi o provocare în datele secvențierii exome, deoarece studiile anterioare au demonstrat că mii de SNV-uri non-sinonime pot fi prezente în oricare dintre Wheeler și colab. (2008). Având în vedere numărul mare de modificări heterozigote non-sinonime, probabilitatea ca vreun SNV heterozigot să fie trecut cu vederea ar putea fi destul de substanțială. Într-adevăr, în timp ce R55C a fost identificat în proiectul de secvențiere a exomului NHLBI, nu există studii care să examineze efectul acestuia. Aceasta ar putea fi o problemă pentru adiponectină, deoarece toate SNV-urile funcționale sunt heterozigote (Jungtrakoon și colab. 2011; Kondo și colab. 2002; Vasseur și colab. 2002).
Deoarece adiponectina joacă un rol important în medierea rezistenței la insulină legate de obezitate (Martin și colab. 2005; Maeda și colab. 2002) în special formele cu greutate moleculară ridicată Pajvani și colab. (2004), identificarea variațiilor funcționale ale adiponectinei ar putea avea implicații clinice substanțiale. Mai exact, dacă aceste variații provoacă hipoadiponectinemie, atunci persoanele cu aceste modificări pot fi sfătuiți să mențină mai agresiv o greutate sănătoasă și o dietă scăzută în carbohidrați, deoarece acești indivizi vor fi cu cel mai mare risc de hiperinsulinemie și T2D. Cu toate acestea, o limitare a acestui studiu este dimensiunea redusă a eșantionului examinat, deci nu este posibil să se stabilească din acest studiu dacă aceste variații pot apărea la alți pacienți și pot duce la aceleași constatări în ceea ce privește oligomerizarea. Prin urmare, este important să se valideze aceste descoperiri biologic și clinic mai întâi. De exemplu, examinarea nivelurilor serice de adiponectină și a oligomerizării la cei 21 de indivizi din Proiectul Exom Sequencing al NHLBI cu o variație R55C ar putea oferi un sprijin suplimentar că această variație este probabil funcțională.
Este important să recunoaștem că, deși această investigație s-a concentrat asupra modificărilor de codificare care pot afecta nivelurile și oligomerizarea adiponectinei, există alte variații în regiunile 5 ’ale genei care sunt asociate cu expresia genei adiponectinei sau nivelurile serice. În special, noi și alții am raportat că variațiile între 10 kb și 12 kb în amonte de ADIPOQ site-ul de început al transcrierii poate modifica nivelurile de adiponectină (Woo și colab. 2006; Heid și colab. 2010; Gupta și colab. 2012), iar polimorfismele promotorului proximal au fost implicate și Laumen și colab. (2009). Deoarece indivizii au fost selectați pentru secvențierea și Western blot pe baza doar fenotipului, mulți adăposteau diverse regiuni promotor, variații sinonime și intronice genotipate anterior în această populație Woo și colab. (2006). Cu toate acestea, într-un set selectat aleatoriu de 54 de persoane din această cohortă (datele nu sunt prezentate), doar un individ a avut HWM la fel de scăzut ca individul nostru cu P32L și nici unul la fel de scăzut ca persoanele noastre cu R55C și MIS. Astfel, este puțin probabil ca aceste alte variații să fie responsabile de adiponectina HWM foarte scăzută prezentă la indivizii noștri cu modificări structurale.
Concluzii
În rezumat, am identificat două noi sinonime ADIPOQ variații folosind o abordare extremă de secvențiere a fenotipului. Persoanele cu aceste variații noi au avut adiponectină scăzută și au prezentat structuri HMW reduse comparativ cu indivizii fără aceste variații. Deși fiecare variație este prezentă în starea heterozigotă, efectele pot fi negative negative. Acest studiu evidențiază utilitatea secvențierii indivizilor cu valori fenotipice extreme pentru a descoperi noi modificări de codificare. Aceste variații pot oferi indicii importante pentru relațiile dintre ADIPOQ variații genetice, distribuția oligomerică a adiponectinei și nivelurile totale de adiponectină circulante. Într-adevăr, studiile viitoare ar putea demonstra că modificările potențiale cauzale rare identificate în acest studiu contribuie la asocierea statistică a variațiilor mai frecvente cu fenotipurile metabolice la populațiile mai mari.
Metode
Protecția subiecților umani
Acest studiu a fost aprobat de Consiliul de revizuire instituțională al Centrului Medical al Spitalului de Copii din Cincinnati. Toți participanții sau părinții/tutorii au acordat consimțământul scris în scris. Subiecții sub 18 ani au dat acordul.
Populația
Proiectarea generală a studiului este o abordare extremă a fenotipului (Figura 3). Pe scurt, au fost selectați 30 de participanți cu niveluri anormale de adiponectină sau fenotipuri specifice ADIPOQ secvențierea; 10 cu cele mai scăzute niveluri de adiponectină (interval 0,6 - 2,5 μg/mL), 10 cu cele mai ridicate niveluri de adiponectină (interval 19,9 - 29,15 μg/ml), 5 diabetice (4 T2D, 1 T1D) și 5 cu rezistență la insulină (interval 127 - 1327 pmol/L insulină). Dintre acești indivizi, 48 la sută erau mici, vârsta medie era de 14,4 ani și existau un număr egal de negri și albi.
Proiectarea studiului a condus la îmbogățirea subiecților cu modificări de sens.
Toți cei 30 de participanți au fost selectați dintr-o cohortă mai mare de 1196 studenți albi și negri care au avut niveluri de adiponectină testate de Martin și colab. (2005). La rândul lor, cei 1196 de participanți au fost selectați aleatoriu dintr-un studiu de 2501 de elevi din districtul școlar Princeton City (Cincinnati, OH), care au fost înscriși de la clasele cinci până la 12 pentru a participa la un studiu școlar despre metabolismul carbohidraților . (2005). Înălțimea și greutatea au fost măsurate și au fost recoltate probe de sânge.
Analize de laborator
Insulina serică de post, glucoza și adiponectina au fost analizate așa cum s-a descris anterior (Martin și colab. 2005; Dolan și colab. 2005). Testele de adiponectină oligomerice (Western Blots și ELISA) au fost efectuate la 12 persoane. Pentru Western Blots, SDS-PAGE a fost realizat conform metodei Laemmli Laemmli (1970). Tamponul de probă pentru condiții de reducere a denaturării căldurii a fost 2% SDS, 50 mM Tris - HCI pH 6,8 și 10% glicerol. Probele au fost incubate peste noapte în tampon de probă la 4 ° C și separate folosind geluri cu gradient SDS-PAGE 4-12% (Jules, Inc., Milford, CT) la o tensiune constantă de 80 V. Proteinele separate prin SDS-PAGE au fost transferate în membranele nitrocelulozice și blocate cu soluție salină tamponată cu Tris cu 0,5% Tween conținând 3% lapte degresat și incubate cu un anticorp policlonal specific pentru om ADIPOQ (0,2 ug/ml; R&D Systems, Minneapolis, MN) peste noapte la temperatura camerei. După o spălare riguroasă, membranele au fost incubate cu IgG asin anti-capră policlonal conjugat HRP (1: 5000; Chemicon, Temecula, CA) timp de 1 oră la temperatura camerei. Benzile au fost detectate folosind reactiv de detectare chemiluminiscent (Pierce, Rockford, IL) și expunerea la film cu raze X (Amersham, Marea Britanie). Benzile LMW, MMW și HMW au fost clasificate ca
75kD, 135-150 kD și
300 kD, respectiv, în concordanță cu rapoartele anterioare (Pajvani și colab. 2004; Waki și colab. 2003); o bandă kD mai mare observată a fost clasificată ca greutate moleculară foarte mare (VHMW). Pentru a explora în continuare profilurile oligomerice, am folosit ALPCO HMW și Total Adiponectin ELISA conform instrucțiunilor producătorului (ALPCO, Salem, NH) Ebinuma și colab. (2006).
Secvențierea
ADN-ul a fost extras din stratul tampon înghețat folosind sistemul Magnesil (Promega Corporation, Madison, WI) automatizat pe un manipulator de margele magnetice KingFisher96 (Thermo Electron, Waltham, MA). Grunduri pentru amplificarea ADIPOQ regiunea de codificare (exonul 2 și capătul 5 ′ al exonului 3, tabelul 4) au fost proiectate folosind ExonPrimer (http://ihg.gsf.de/ihg/ExonPrimer.html). Fragmentul de 1.954 bp a fost amplificat din 50 ng de ADN genomic utilizând sistemul PCR Gold AmpliTaq (Applied Biosystems, Foster City, CA) modificat din condiții standard prin creșterea temperaturii de recoacere la 65 ° C și a timpului de extindere la 3 minute. Primerii și nucleotidele PCR au fost îndepărtați enzimatic din amplicon înainte de secvențierea directă utilizând ExoSAP-IT (GE Healthcare, Fairfield, CT). Un total de 4 reacții de secvențiere au fost efectuate pentru a obține secvențierea bidirecțională a întregii regiuni de codificare (primeri din Tabelul 4) folosind kitul BigDye v3.1 Terminator pe un analizor ADN 3730xl (Applied Biosystems). Analiza SNV a fost efectuată utilizând software-ul Mutation Surveyor v2.61 (SoftGenetics, State College, PA).
Persoanele cu modificări de codificare non-sinonime au fost potrivite în funcție de vârstă (± 6 luni), stadiul pubertății, sex și rasă la indivizi din cohorta mai mare. Un control (Match-A) a fost ales cu scor IM și insulină similar, având în vedere potrivirea demografică. S-a ales un al doilea control (Match-B), care se potrivește pe variabilele demografice și scorul Z IMC, dar diferit pe insulină. Controalele potrivite au fost secvențiate pentru a se asigura că nu sunt adăpostite ADIPOQ modificări de codificare.
analize statistice
Scorul Z IMC a fost calculat din graficele de creștere CDC. Rezistența la insulină a fost definită ca niveluri de insulină> 95% ile de indivizi slabi în cohorta totală de 2501 studenți cu aceeași etapă de rasă, sex și pubertate Dolan și colab. (2005). Pentru a determina dacă indivizii cu ADIPOQ modificările de codificare au prezentat fenotipuri extreme, am calculat percentilele 5 și 95 pentru vârstă, adiponectină, insulină, glucoză și IMC scor Z, folosind cohorta noastră de 1196 studenți Martin și colab. (2005). Pentru a compara frecvența detectării SNV-urilor non-sinonime cu o bază de date publică (NHLBI Exome Sequencing Project), am folosit Fisher Exact Test.
- Măsurarea automată rapidă a distribuției grăsimii corporale din RMN-ul întregului corp American Journal of
- Retailerul rus de produse alimentare X5 Retail deschide un centru de distribuție în Yaroslavl
- Semnalizare translațională PLOS ONE, expresie genică atrogenă și miogenică în timpul descărcării și
- Reddit - AsianBeauty - Masajul facial poate schimba efectiv distribuția grăsimilor de pe față
- Cârnați tradiționali portughezi de diferite tipuri, compoziție nutrițională și tendințe noi -