Braulio J Soto-Cerda
1 Departamentul de Științe ale Plantelor, Universitatea din Manitoba, 66 Dafoe Road, Winnipeg, MB R3T 2N2, Canada
2 Cereal Research Center, Agricultură și Agroalimentare Canada, 195 Dafoe Rd, Winnipeg, MB R3T 2M9, Canada
4 Adresa actuală: Agriaquaculture Nutritional Genomic Center, CGNA, Genomics and Bioinformatics United Kingdom, Km 10 Camino Cajón-Vilcún, INIA Temuco, Chile
Axel Diederichsen
3 Resurse genetice vegetale din Canada, Agricultură și Agroalimentare Canada, 107 Science Place, Saskatchewan, SK S7N 0X2, Canada
Raja Ragupathy
1 Departamentul de Științe ale Plantelor, Universitatea din Manitoba, 66 Dafoe Road, Winnipeg, MB R3T 2N2, Canada
2 Cereal Research Center, Agricultură și Agroalimentare Canada, 195 Dafoe Rd, Winnipeg, MB R3T 2M9, Canada
Sylvie Cloutier
1 Departamentul de Științe ale Plantelor, Universitatea din Manitoba, 66 Dafoe Road, Winnipeg, MB R3T 2N2, Canada
2 Cereal Research Center, Agricultură și Agroalimentare Canada, 195 Dafoe Rd, Winnipeg, MB R3T 2M9, Canada
Date asociate
Abstract
fundal
Inul este apreciat pentru fibre, ulei de semințe și nutraceutice. Recent, industria fibrelor a investit în dezvoltarea produselor fabricate din tulpini de in, făcându-l o cultură cu dublu scop. Direcționarea simultană a regiunilor genomice care controlează trăsăturile de calitate a semințelor și a fibrelor ar putea permite dezvoltarea de soiuri cu scop dublu. Cu toate acestea, diversitatea genetică, structura populației și modelele de dezechilibru de legătură (LD) necesare pentru cartografierea asocierii (AM) nu au fost încă evaluate în in, deoarece resursele genomice au fost dezvoltate abia recent. Am caracterizat 407 accesiuni de in distribuite la nivel global folosind 448 de markeri microsateliți. Datele au fost analizate pentru a evalua adecvarea acestei colecții de bază pentru AM. Scanările genomice pentru a identifica genele candidate selectate în timpul procesului de reproducere divergent al inului și semințelor de in au fost efectuate folosind întreaga secvență a inimii de pușcă a genomului.
Rezultate
Analiza combinată a structurii genetice a atribuit toate aderările la două grupuri majore cu șase subgrupuri. Diferențierea populației a fost slabă între principalele grupuri (FST = 0,094) și pentru majoritatea comparațiilor perechi între subgrupuri. Analiza coancesterii moleculare a indicat o relație slabă (medie = 0,287) pentru majoritatea perechilor individuale. Diversitatea genetică abundentă a fost observată în totalul panoului (5,32 alele per locus), iar unele subgrupuri au prezentat o proporție mare de alele private. LD mediu la nivelul genomului (r 2) a fost de 0,036, cu o descompunere relativ rapidă de 1,5 cM. Scanările genomice dintre fibrele de in și semințele de in au identificat gene candidate implicate în biogeneza/modificarea peretelui celular, identitatea xilemului și biosinteza acizilor grași congruenți cu genele identificate anterior în in și alte specii de plante.
Concluzii
Pe baza diversității genetice abundente, a structurii și a legăturii slabe a populației și a degradării relativ rapide a LD, am concluzionat că această colecție de bază este potrivită pentru studiile AM care vizează multiple trăsături agronomice și de calitate care vizează îmbunătățirea inului ca o adevărată cultură cu dublu scop. Scanările noastre genomice oferă primele informații despre regiunile candidate afectate de selecția divergentă în in. În combinație cu AM, scanările genomice au capacitatea de a crește puterea de a detecta loci care influențează trăsături complexe.
fundal
Inul (Linum usitatissimum L.) este o specie anuală, auto-polenizată, cu o dimensiune a genomului de
Evaluările inițiale ale diversității la in au fost efectuate utilizând parametri morfologici [9-12] și izozime [13,14]. În ultimii ani, sistemele de markeri moleculari, cum ar fi ADN-ul polimorf amplificat aleatoriu (RAPD), polimorfismul de lungime a fragmentului amplificat (AFLP), repetarea secvenței inter-simple (ISSR), repetarea secvenței simple (SSR) și polimorfismul amplificat inter-retrotransposon (IRAP) au au fost folosite pentru a măsura variația genetică și relațiile din soiuri și rase de in [15-29]. Cu toate acestea, majoritatea acestor studii anterioare au evaluat fie puțini loci markeri, fie puține genotipuri.
Băncile mondiale de gene stochează aproximativ 48.000 de accesiuni de germoplasmă de in [30]. În Canada, o colecție mondială de aproximativ 3.500 de accesiuni de in cultivat este întreținută de Plant Gene Resources of Canada (PGRC). Această colecție a fost desfășurată în mod tradițional în reproducerea inului printr-o varietate de strategii convenționale [3]. În 2009, proiectul Total Utilization Flax Genomics (TUFGEN; http://www.tufgen.ca) a fost inițiat în Canada pentru a genera resurse de genomică pentru in și pentru a le aplica la o serie de trăsături în scopul final al îmbunătățirii inului. Proiectul TUFGEN a dezvoltat numeroase resurse de genomică, inclusiv markeri moleculari [23,29,31], hărți genetice [32,33], o hartă fizică și secvențe de capăt ale cromozomilor artificiali bacterieni [1], etichete de secvență exprimate [34] și pușcă cu genom întreg secvența [35]. Pentru a profita de aceste instrumente, a fost asamblată o colecție centrală de 407 accesorii de in care capturează diversitatea fenotipică a colecției PGRC.
Lociile cantitative ale trăsăturilor (QTL) și maparea asocierii (AM) sunt abordări complementare pentru identificarea asocierii marker-trăsături. Primul folosește populații de cartografiere biparentale pentru a monitoriza co-segregarea QTL și a locurilor marker. A doua folosește colecții de germoplasmă pentru a identifica corelațiile markerului QTL bazate pe LD [36]. Analiza QTL are o rezoluție limitată a cartografierii datorită acumulării de puține evenimente de meioză într-o singură cruce, dar nu este afectată de structura populației care poate fi o sursă de asociere falsă în AM. În schimb, AM poate obține o rezoluție mai mare de cartografiere prin un număr mare de evenimente istorice de recombinare în colecțiile de germoplasmă. Un panou de asociere ideal ar trebui să găzduiască cea mai largă diversitate genetică, deoarece aceasta este adesea corelată cu o descompunere rapidă a LD necesară pentru a rezolva variațiile complexe ale trăsăturilor la o singură genă sau nucleotidă [37]. Structura populației nule sau slabe și un nivel scăzut de legătură între indivizii din colecția de germoplasmă sunt de asemenea de dorit. Astfel, diversitatea genetică, structura populației, relația familială și modelele LD trebuie să fie evaluate înainte de analizele AM pentru a exploata pe deplin avantajele lor pentru îmbunătățirea genetică a inului.
În acest studiu am genotipat 407 accesiuni de in folosind 448 loci microsatelite. Scopul general a fost de a evalua utilitatea acestei colecții de miez de in pentru studiile AM. Obiectivele noastre specifice au fost: (1) investigarea diversității genetice; (2) să estimeze nivelurile structurii populației și să evalueze relația familială; (3) pentru a detecta tiparele LD; și (4) identificarea regiunilor genomice non-neutre care pot sta la baza selecției divergente între tipurile de fibre și semințe de in.
Rezultate
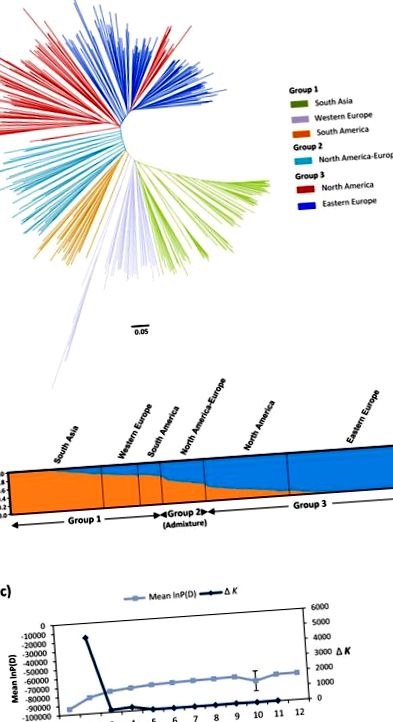
Analiza filogenetică
Distribuția estimărilor periferice moleculare și a dezechilibrului de legătură. (A) Estimări globale de pereche moleculară pe perechi a 407 accesiuni de in din colecția de bază. Sunt afișate numai valorile de rudenie cuprinse între 0 și 0,5. (b) Graficul de împrăștiere a decăderii LD (r 2) față de distanțele genetice (cM) pentru perechi de SSR legate între cele 15 grupuri de legătură. Panoul interior prezintă o vedere detaliată a decăderii LD pentru markerii aflați la 5 cM. Curbele de descompunere au fost trasate conform lui Breseghello și Sorells [75]. Linia albastră reprezintă nivelul pragului de semnificație (r 2 = 0,1). Linia roșie reprezintă LD medie la nivelul genomului de markeri legați. (c) Estimări de coacesterie moleculară pereche [72] în fiecare dintre cele șase subgrupuri. Valorile diagonale corespund coancesterii moleculare intra-grup. (d) Curbele medii ale descompunerii LD la nivelul genomului pentru markerii legați din fiecare dintre cele șase subgrupuri.
Diversitate genetică
În colecția de miez, cei 414 microsateliți neutri reținuți au detectat 2202 alele (Na) (medie = 5,32/locus), din care 1187 (54%) au avut un MAF 1). Parametrii Na, Rs, ∏, Ra și PIC în G1 au fost superiori celor din G3, chiar dacă dimensiunea populației G1 a fost cu 25% mai mică decât G3. Parametrii Ho și FIS de-a lungul colecției de bază, principalele grupuri și subgrupuri sunt în concordanță cu natura predominant auto-polenizată a speciei.
tabelul 1
Parametrii de diversitate genetică a colecției de bază pentru cele două grupuri majore (G1 și G3), grupul amestecat (G2) și sub-grupuri
| Colecția de bază | 407 | 0,427 | 0,023 | 2202 | 5,68 | - | 1187 | 0,946 | 100 | 0,374 |
| Grupa 1 | 153 | 0,418 | 0,023 | 1978 | 4.37 | 547 | 925 | 0,944 | 99,8 | 0,361 |
| Asia de Sud | 92 | 0,348 | 0,020 | 1510 | 2,85 | 116 | 542 | 0,931 | 95,9 | 0,305 |
| Europa de Vest | 37 | 0,488 | 0,017 | 1608 | 3,44 | 246 | 418 | 0,961 | 97.1 | 0,393 |
| America de Sud | 24 | 0,395 | 0,047 | 1135 | 2,70 | 27 | 186 | 0,878 | 91.3 | 0,332 |
| Grupa 2 | ||||||||||
| Nord Amer./Eur. | 43 | 0,41 | 0,023 | 1341 | 2,91 | 32 | 324 | 0,933 | 96,4 | 0,352 |
| Grupa 3 | 211 | 0,356 | 0,022 | 1613 | 3,44 | 183 | 683 | 0,933 | 99.1 | 0,332 |
| America de Nord | 95 | 0,378 | 0,028 | 1362 | 2,69 | 73 | 424 | 0,932 | 98,6 | 0,334 |
| Europa de Est | 116 | 0,300 | 0,020 | 1487 | 2,55 | 45 | 642 | 0,927 | 95,7 | 0,265 |
1 Număr de aderări.
2 Diversitate de gene nepărtinitoare.
3 Heterozigoza observată.
4 Numărul de alele.
5 bogăția alelică și 6 numărul de alele private estimate pe un eșantion de dimensiuni echilibrate utilizând metoda rarefacției [66].
7 Alele rare 8 Coeficient de consangvinizare.
9 Conținutul informațiilor despre polimorfism.
Dezechilibru de legătură
masa 2
Dezechilibru de legătură în colecția de bază pentru cele două grupuri majore (G1 și G3), grupul amestecat (G2) și cele șase sub-grupuri
- Caracterizarea speciilor de bacil utilizate pentru bacterioterapia orală și bacterioprofilaxia
- Funcționează Wonder Core Smart așa cum se vede la televizor
- Fapte de sănătate ale lui Clayton Semințe de in
- Urmărirea tulburărilor genetice Young Zoya este acum mai ușoară, mai fericită după lupta împotriva obezității - Oglinda Mumbai
- Ezekiel 4 9 Recenzie de pâine de cereale încolțită de in - Pur și simplu Stacie