Lili Yang 1.2 #, Yaoyao Bian 3 #, Zhengjun Li 4 #, Yan Yan 5, Junyi Li 1, Wenlin Li 2, Li Zeng 1.2
Contribuții: (I) Concepție și proiectare: L Yang, Y Bian; (II) Sprijin administrativ: Nici unul; (III) Furnizarea de materiale de studiu sau de pacienți: Nici unul; (IV) Colectarea și asamblarea datelor: Y Bian; (V) Analiza și interpretarea datelor: Z Li, J Li; (VI) Scrierea manuscriselor: Toți autorii; (VII) Aprobarea finală a manuscrisului: Toți autorii.
# Acești autori au contribuit în mod egal la această lucrare.
Fundal: Colita ulcerativă (UC) este o boală inflamatorie cronică, recidivantă și nespecifică, care implică diferite gene și căi în patogeneza lor. Dovezi în creștere au arătat că microARN-urile (miARN-uri) acționează ca regulatori cheie post-transcripționali ai expresiei genice în UC. Acest studiu actual a vizat identificarea miARN-urilor cheie, a genelor potențiale țintă și a căilor relevante implicate în UC pentru a-și descoperi mecanismele moleculare de bază utilizând analiza bioinformatică.
Metode: Profilurile de expresie mARN și miARN au fost recuperate și descărcate din baza de date Genn Expression Omnibus (GEO). Genele exprimate diferențial (DEG) și miARN (DEMI) au fost obținute utilizând pachetul software R.
Rezultate: Au fost obținute un total de 79 DEG și 47 DEMI. Și un grup de miARN și mARN-urile lor țintă au fost identificate. A arătat că miR-1231 poate fi un regulator cheie pentru DUOX2 și TFF1. CCL11 poate fi potențial vizat de miR-625. MMP1 poate juca roluri vitale în dezvoltarea UC prin reglarea căii de semnalizare miR-1228/PPAR. În plus, am validat cel mai semnificativ miARN exprimat în sus/în jos (miR-92b, miR-625) și doi dintre mARN-urile lor corespunzătoare (AQP8 și TAGAP, CCL11 și CHI3L1) în țesuturile colonului din modelele UC preliminar. Rezultatele au fost în concordanță cu analiza microarray.
Concluzii: Aceste descoperiri pot oferi noi perspective asupra reprezentării mecanismelor cheie asociate cu dezvoltarea UC.
Cuvinte cheie: Colita ulcerativă (UC); microARN (miARN); gene exprimate diferențial (DEG); analiza bioinformatică; analiza îmbogățirii funcționale
Trimis 06 martie 2019. Acceptat pentru publicare 03 iunie 2019.
Introducere
Colita ulcerativă (UC), un subtip de boală inflamatorie intestinală (IBD), este o boală inflamatorie cronică, recidivantă și nespecifică, care se limitează la mucoasa și submucoasa rectului sau colonului. Se caracterizează prin două perioade de boală activă și remisie. Manifestările clinice tipice sunt diareea, scaunul purulent și durerea abdominală (1).
UC afectează milioane de oameni din întreaga lume. Cele mai mari incidențe ale UC în America de Nord și Europa de Nord au fost de 6-15,6 și respectiv 10-20,3 cazuri la 100.000 anual (2). O analiză recentă a 44 de studii care a inclus 31.287 de pacienți asiatici a constatat o rată de prevalență de 0,85% a UC (3). În plus, pacienții care suferă de UC prezintă un risc ridicat de a dezvolta cancer colorectal (CRC) (4). UC a adus o povară personală și socială semnificativă. În plus, UC activ poate reduce calitatea vieții fizice și mentale și poate crește stresul psihologic.
În acest studiu, am preluat seturi de date de microarrays de expresie mARN și miARN de la Omnibusul de Expresie Genică (GEO) și am identificat un grup de miARN cheie și gene potențial țintă implicate în UC utilizând analiza bioinformatică. În plus, miARN-urile cele mai semnificative exprimate și genele lor țintă au fost selectate și efectuate pentru validare preliminară prin qPCR în timp real (qRT-PCR). Studiul a urmărit să sugereze semnături miARN utile pentru detectarea și diagnosticarea activă a UC, precum și să exploreze patogeneza subiacentă prin identificarea potențialei ARNm direcționate către miARN la nivel molecular.
Metode
Criterii de colectare și includere a studiilor
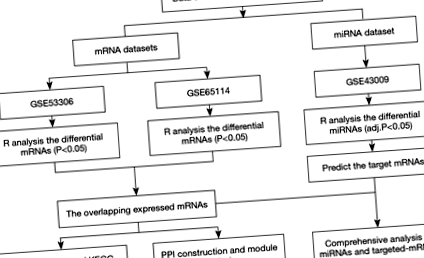
Seturile de date ale profilului de expresie microarray mRNA au fost recuperate și descărcate din baza de date GEO (disponibilă online: http://www.ncbi.nlm.nih.gov/geo) prin căutarea următoarelor cuvinte cheie: „ARN”, „colită ulcerativă activă”, și „Homo sapiens” (organism). Criteriile de includere au fost după cum urmează: (I) țesuturi de colon de la pacienți adulți cu UC activă (nu celule); (II) probe din grupul UC fără a primi intervenții sau tratamente; și (III) ambele numere de UC și eșantionul de control al sănătății ≥12. În plus, seturile de date ale profilului miARN au fost căutate utilizând cuvintele cheie: „miARN”, „colită ulcerativă activă” și „Homo sapiens” (organism) în conformitate cu următoarele criterii: (I) țesuturile colonului de la pacienții UC activi (nu celulele) și (II) probe din grupul UC fără a primi intervenții sau tratamente. După screening, au fost selectate două seturi de date de expresie mARN [GSE53306 (16) și GSE65114] și a fost obținut un set de date de expresie miARN (GSE43009) pentru analiză. Fluxul de lucru al procesării și analizei datelor este prezentat în Figura 1.
Date microarray
În acest studiu, platforma pentru GSE53306 s-a bazat pe GPL14951 Illumina HumanHT-12 WG-DASL V4.0 R2 beadchip de expresie, care a constat din 16 probe active UC și 12 controale. Platforma pentru GSE65114 a fost matricea GPL16686 (HuGene-2_0-st) Affymetrix genă umană 2.0 ST, care a inclus 16 probe active UC și 12 controale. Platforma pentru GSE43009 s-a bazat pe matrice GPL16384 Affymetrix multispecie miRNA-3, care a constat din cinci controale și cinci probe UC.
Prelucrarea datelor și gene exprimate diferențial (DEG)/identificare diferențială a miARN (DEMI)
Datele brute au fost descărcate din baza de date GEO și apoi normalizate și standardizate utilizând pachetul software R. Analiza expresiei diferențiale genice a fost efectuată prin pachetele limma din pachetul Bioconductor (17) (disponibil online: http://www.bioconductor.org/). Hărțile de căldură ale celor două seturi de date mRNA au fost mapate utilizând pachetul gplots din R pentru a vizualiza valorile de expresie ale genelor din diferitele probe. Când am selectat DEG-urile, P 1 a fost considerat ca fiind valorile limită, unde FC este o schimbare de ori. DEMI semnificative au fost examinate prin întâlnirea ambilor adj. P 1. Instrumentul online Venny (disponibil online: http://bioinfogp.cnb.csic.es/tools/venny) a fost utilizat pentru a identifica DEG-urile din cele două seturi de date ale ARNm. DEMI-urile identificate au fost păstrate pentru analize bioinformatice ulterioare.
Analiza funcțională de îmbogățire a DEG-urilor
Analiza de îmbogățire a ontologiei genetice (GO), care este utilizată pentru adnotarea genelor, a fost efectuată pentru a determina funcțiile reglementate semnificativ, adică procesele biologice (TA), componenta celulară (CC) și funcția moleculară (MF). Analiza de îmbogățire a Enciclopediei Kyoto a Genelor și Genomelor (KEGG) a fost efectuată pentru prezentarea analizei sistematice, adnotării și vizualizării funcțiilor genetice. Atât îmbogățirea GO, cât și analiza căii KEGG au fost realizate folosind baza de date pentru adnotare, vizualizare și descoperire integrată (DAVID) (disponibilă online: http://david.abcc.ncifcrf.gov/) pentru a identifica semnificația biologică a genelor, atunci când P 0,4 definit ca criteriu de tăiere. Rețelele de reglementare integrate au fost apoi vizualizate de Cytoscape (18). În cele din urmă, plug-in-ul Molecular Complex Detection (MCODE) a fost aplicat pentru ecranarea modulelor rețelei PPI.
Predicția țintelor miARN
Țintele relevante ale miARN au fost prezise folosind miRWalk (disponibil online: http://zmf.umm.uni-heidelberg.de/apps/zmf/mirwalk2/), care este un atlas cuprinzător al interacțiunilor predictive și validate miARN-țintă. Țintele potențiale ale miARN au fost identificate de cel puțin patru programe.
Animale și design experimental
Douăzeci de șobolani masculi SD (180 ± 20 g) în vârstă de 8-10 săptămâni au fost cumpărați de la ferma de reproducere a animalelor experimentale Qinglongshan (Nanjing, China). Animalele au fost adăpostite într-o stare standard, cu temperatura ambiantă de 22 ± 2 ° C la 12 h lumină/întuneric. Șobolanii cu acces nelimitat la șobolanul standard au fost repartizați aleatoriu în două grupuri (zece șobolani pe grup). Șobolanii cu colită acută au fost induși prin administrarea de apă potabilă conținând 3,5% (g/v) dextran sulfat de sodiu (DSS) (Sigma-Aldrich, SUA), în timp ce controalelor li s-a dat apă potabilă fără DSS. După 10 zile, toți șobolanii au fost sacrificați sub anestezie cu izofluran și țesuturile colonului au fost recoltate pentru analiză ulterioară. Toate protocoalele efectuate pentru animale au fost conforme cu Ghidul chinez de cazare și îngrijire pentru animale formulat și sub aprobarea Universității de Medicină Chineză din Nanjing.
Extracția ARN și qRT-PCR
Conform instrucțiunilor producătorului, ARN-ul total al țesuturilor colonului în ambele grupuri UC și martori au fost extrase folosind reactivul TRIzol (Invitrogen, California, SUA). Primerii au fost obținuți din Genscript, iar secvențele au fost prezentate în Tabelul S1. Expresia mARN și miARN a fost normalizată față de expresia GAPDH și respectiv U6.
Colorarea hematoxilinei și eozinei (HE)
Țesuturile colonului au fost tăiate într-o secțiune de 4 m grosime după fixare, deshidratare și încastrare și apoi colorate cu hematoxilină și eozină. Trei secțiuni aleatorii ale fiecărui țesut au fost realizate sub microscop (Leica, germană).
Analize statistice
Analizele statistice au fost efectuate utilizând software-ul SPSS 19.0. Toate datele au fost exprimate ca medie ± deviație standard (SD) și analizate folosind Graph Pad Prism (Versiunea 5.0, Inc., CA, SUA). Comparații statistice în cadrul a două grupuri au fost făcute prin teste t Student nepereche. Și valoarea P mai mică de 0,05 a fost considerată semnificativă.
Rezultate
Identificarea DEG-urilor
Cele două seturi de date microarray GSE53306 și GSE65114 au fost normalizate, așa cum se arată în Figura 2. Un total de 1.052 DEG-uri au fost selectate din setul de date GSE53306. În plus, 296 DEG-uri au fost identificate din setul de date GSE65114. Hărțile de căldură ierarhice în cluster ale DEG-urilor din ambele seturi de date sunt prezentate în Figura 3. Și 79 de gene au fost exprimate diferențial în ambele două seturi de date.
Analize funcționale și de îmbogățire a căilor de DEG
Cele 79 de gene suprapuse de mai sus au fost încărcate în instrumentul de analiză online DAVID pentru atribuirea funcțională și îmbogățirea căilor cu o valoare P 0,4. Am găsit mai multe gene hub, cum ar fi inhibitorul metalopeptidazei 1 (TIMP1), ligandul chemokinei CXC motiv 10 (CXCL10), ligandul chemokinei CXC motiv 13 (CXCL13), ligandul chimokinei 11 motiv CC (CCL11), ligandul chemokinei motiv CC 19 (CCL11) și selectați L (VÂNZARE).
Ulterior, cele 55 de perechi PPI obținute au fost analizate folosind Cytoscape pentru a descrie relația complexă. Mai mult, au fost identificate două clustere din plug-in-ul DCOME, care consta din nouă gene (Figura 5B), SELL, CCL19, CXCL13, CXCL10, CCL11, subunitatea de reglare ribonucleotid reductază M2 (RRM2), kinaza de legare PDZ (PBK), centrosomală proteina 55 (CEP55) și interactorul receptorului hormonului tiroidian 13 (TRIP13). În cele din urmă, am constatat că aceste gene au fost îmbogățite în răspunsul imun de către DAVID.
Predicția DEMI și identificarea genelor potențiale țintă
Setul de date de profilare GSE43009 a fost utilizat pentru a identifica DEMI-urile. După preprocesarea datelor, am eliminat un grup din eșantionul UC și am controlat eșantionul din cauza clusterului ierarhic slab. Au fost identificate în total 47 de DEMI. Apoi, prin utilizarea bazei de date miRWalk, s-au obținut țintele prezise ale miARN-urilor. ARNm-urile suprapuse prezise de miRWalk și DEG-urile identificate mai sus sunt prezentate în tabelul 3. Am constatat că miR-92b și miR-625 au fost cele mai semnificative miARN. PTGIS a fost prezis ca țintă potențială a patru miARN, și anume, miR-1228, miR-1268, miR-1231 și miR-92b. DES a fost identificat ca o țintă potențială a trei miARN, și anume, miR-939, miR-1268 și miR-1226. UNC13D a fost găsit ca o potențială țintă de miR-939, miR-1268 și miR-1908. Mai mult, șapte gene (inclusiv MMP10, DPP10 și PCK1) au fost potențial vizate de miR-92b, în timp ce șapte ARNm (inclusiv CCL11) au fost potențial vizate de miR-625.
Verificarea miARN și a mARN-urilor țintă prin qRT-PCR
Pentru a demonstra miARN-urile și mARN-urile țintă pe care le-am identificat, am ales cele mai semnificative miARN exprimate în sus/în jos (miR-92b și miR-625) și am selectat două dintre mARN-urile lor țintă corespunzătoare (AQP8 și TAGAP, CCL11 și CHI3L1) pe baza rezultatelor analizei bioinformatice și a literaturilor anterioare pentru validarea în țesuturile colonului pe modelele UC de rozătoare. Colorarea HE a UC și controlul indus de DSS (Figura 6) a sugerat pregătirea modelului cu succes. Rezultatele qRT-PCR au indicat că miARN-urile și mARN-urile corespunzătoare au fost exprimate semnificativ (P
Concluzii
În studiul de față, un total de 79 DEG și 47 DEMI au fost identificate din seturile de date GEO, care ar putea oferi dovezi inițiale că aceste gene și miARN pot servi ca semnături potențiale legate de UC activă. Geneza UC este un proces extrem de complex în timpul căruia apar multe modificări genetice și epigenetice ale genelor conducătoare.
Dintre genele hub pe care le-am identificat, CXCL10 a fost evidențiat ca fiind cel mai înalt grad de gene de conectivitate. CXCL10, precum și CXCL13 au fost raportate o creștere semnificativă la pacienții cu IBD (19). TIMP1 este un membru al familiei de gene TIMP și codifică metaloproteinazele matriciale (MMP), care sunt factori cheie ai celulelor T mediată de leziuni tisulare. Ambele gene sunt implicate în răspunsul inflamator după deteriorarea țesutului și repararea în IBD. TIMP1 a fost detectat în războiul cu boala asemănătoare Crohn (20). Un alt studiu (21) bazat pe șoareci knock-out a arătat că deficitul de TIMP1 poate provoca o expresie ridicată a genelor legate de imunitate. CCL19, ca membru al citokinelor CC, se exprimă în mod semnificativ în boala Crohn (CD) și poate duce în mod normal la microambientul chemokinelor (22). În lumina rezultatelor de mai sus, am presupus în continuare că aceste gene pot reprezenta biomarkeri candidați pentru UC.
Cu toate acestea, prezentul studiu vine cu unele limitări. În primul rând, rezultatele au fost obținute dintr-o bază de date publică de microarray GEO și platformele de analiză a trei seturi de date GSE nu au fost uniforme. În al doilea rând, probele au fost limită, ceea ce poate cauza fiabilitatea concluziei noastre. Sunt necesare studii suplimentare cu mai multe eșantioane și o platformă de detectare tehnologică unificată pentru a confirma rezultatele noastre.
Luate împreună, studiul nostru actual a folosit o analiză bioinformatică cuprinzătoare pentru a determina expresia mARN și miARN între UC activ și control. Un grup de miARN și genele lor țintă au fost identificate și mai multe dintre ele au fost confirmate preliminar pe modelul rozătoarelor, care pot servi ca potențiali biomarkeri legați de UC. În plus, am găsit mai multe funcții și căi genetice importante, care ne pot ajuta să înțelegem mecanismele moleculare ale UC. Cu toate acestea, studii experimentale și funcționale suplimentare sunt justificate pentru a determina rolul și mecanismele exacte ale UC.
Mulțumiri
Finanțare: Acest studiu a fost sprijinit de subvenții de la Fundația Națională pentru Științe Naturale din China (Grant nr. 81704084, 81673982 și 81603529), Proiectele de știință și tehnologie ale Jiangsu Provincial Bureau of Traditional Chinese Medicine (YB2017002 și YB2015002), Științele naturale Fundația instituțiilor de învățământ superior din Jiangsu (16KJB360002), Programul de cercetare postuniversitară și programul de inovare a practicii din provincia Jiangsu (KYCX18_1541) și sponsorizat de proiectul Qing Lan, proiectul de finanțare a disciplinei de avantaj pentru medicina chineză și Consiliul de bursă China (CSC).
Notă de subsol
Conflictele de interese: Autorii nu au conflicte de interese de declarat.
Declarație etică: Autorii sunt responsabili pentru toate aspectele lucrării, asigurându-se că întrebările legate de acuratețea sau integritatea oricărei părți a lucrării sunt investigate și rezolvate în mod corespunzător.
- IBD Crohn; s Boala Colita Ulcerativa; Dieta WFPB partea 5 din 5; Dieta alimentară integrală pe bază de plante
- Cum mi-am luat colita ulcerativă sub control
- Identificarea biomarkerilor salivari și plasmatici pentru obezitate la copii de către ne-vizați
- Fibra dietetică este bună pentru pacienții cu Crohn; s Boala sau colita ulcerativă AGA Reviste Blog
- Identificarea genelor și căilor cheie asociate cu obezitatea la copii