Soonok Kim
1 Divizia de evaluare a resurselor biologice și genetice, Institutul Național de Resurse Biologice, Incheon, 22689 Republica Coreea
Yun Sung Cho
2 Institutul de Genomică, Institutul Național de Știință și Tehnologie Ulsan (UNIST), Ulsan, 44919 Republica Coreea
3 Departamentul de Inginerie Biomedică, Școala de Științe ale Vieții, Institutul Național de Știință și Tehnologie Ulsan (UNIST), Ulsan, 44919 Republica Coreea
4 Personal Genomics Institute, Genome Research Foundation, Cheongju, 28160 Republica Coreea
Hak-Min Kim
2 Institutul de Genomică, Institutul Național de Știință și Tehnologie Ulsan (UNIST), Ulsan, 44919 Republica Coreea
3 Departamentul de Inginerie Biomedică, Școala de Științe ale Vieții, Institutul Național de Știință și Tehnologie Ulsan (UNIST), Ulsan, 44919 Republica Coreea
Oksung Chung
4 Personal Genomics Institute, Genome Research Foundation, Cheongju, 28160 Republica Coreea
Hyunho Kim
5 Geromics, Ulsan, 44919 Republica Coreea
Sungwoong Jho
4 Personal Genomics Institute, Genome Research Foundation, Cheongju, 28160 Republica Coreea
Hong Seomun
6 Divizia de resurse animale, Institutul Național de Resurse Biologice, Incheon, 22689 Republica Coreea
Jeongho Kim
7 Cheongju Zoo, Cheongju, 28311 Republica Coreea
Woo Young Bang
1 Divizia de evaluare a resurselor biologice și genetice, Institutul Național de Resurse Biologice, Incheon, 22689 Republica Coreea
Changmu Kim
1 Divizia de evaluare a resurselor biologice și genetice, Institutul Național de Resurse Biologice, Incheon, 22689 Republica Coreea
Junghwa An
6 Divizia de resurse animale, Institutul Național de Resurse Biologice, Incheon, 22689 Republica Coreea
Chang Hwan Bae
1 Divizia de evaluare a resurselor biologice și genetice, Institutul Național de Resurse Biologice, Incheon, 22689 Republica Coreea
Youngjune Bhak
2 Institutul de Genomică, Institutul Național de Știință și Tehnologie Ulsan (UNIST), Ulsan, 44919 Republica Coreea
Sungwon Jeon
2 Institutul de Genomică, Institutul Național de Știință și Tehnologie Ulsan (UNIST), Ulsan, 44919 Republica Coreea
3 Departamentul de Inginerie Biomedică, Școala de Științe ale Vieții, Institutul Național de Știință și Tehnologie Ulsan (UNIST), Ulsan, 44919 Republica Coreea
Hyejun Yoon
2 Institutul de Genomică, Institutul Național de Știință și Tehnologie Ulsan (UNIST), Ulsan, 44919 Republica Coreea
3 Departamentul de Inginerie Biomedică, Școala de Științe ale Vieții, Institutul Național de Știință și Tehnologie Ulsan (UNIST), Ulsan, 44919 Republica Coreea
Yumi Kim
2 Institutul de Genomică, Institutul Național de Știință și Tehnologie Ulsan (UNIST), Ulsan, 44919 Republica Coreea
JeHoon iun
4 Personal Genomics Institute, Genome Research Foundation, Cheongju, 28160 Republica Coreea
5 Geromics, Ulsan, 44919 Republica Coreea
HyeJin Lee
4 Personal Genomics Institute, Genome Research Foundation, Cheongju, 28160 Republica Coreea
5 Geromics, Ulsan, 44919 Republica Coreea
Suan Cho
4 Personal Genomics Institute, Genome Research Foundation, Cheongju, 28160 Republica Coreea
5 Geromics, Ulsan, 44919 Republica Coreea
Olga Uphyrkina
8 Institutul de Biologie și Știința Solului, Filiala Orientului Îndepărtat a Academiei Ruse de Științe, Vladivostok, 690022 Rusia
Aleksey Kostyria
8 Institutul de Biologie și Știința Solului, Filiala Orientului Îndepărtat a Academiei Ruse de Științe, Vladivostok, 690022 Rusia
John Goodrich
9 Panthera, New York, NY 10018 SUA
Dale Miquelle
10 Wildlife Conservation Society, 2300 Southern Boulevard, Bronx, NY 10460 SUA
11 Departamentul de ecologie, Universitatea Federală din Orientul Îndepărtat, Ayaks, Islanda Rusă, Vladivostok, 690950 Rusia
Melody Roelke
Programul 12 Laborator de Științe Animale, Leidos Biomedical Research Inc., Laboratorul Național Frederick, Frederick, MD 21702 SUA
John Lewis
13 International Zoo Veterinary Group (UK) IZVG LLP, Station House, Parkwood Street, Keighley, BD21 4NQ UK
Andrey Yurchenko
14 Theodosius Dobzhansky Center for Genome Bioinformatics, St. Universitatea de Stat St. Petersburg, St. Petersburg, 199004 Rusia
Anton Bankevich
15 Centrul pentru Biotehnologie Algoritmică, Institutul de Biomedicină Translațională, St. Universitatea de Stat St. Petersburg, St. Petersburg, 199034 Rusia
Juok Cho
16 Broad Institute of MIT și Harvard, Cambridge, MA 02142 SUA
Semin Lee
2 Institutul de Genomică, Institutul Național de Știință și Tehnologie Ulsan (UNIST), Ulsan, 44919 Republica Coreea
3 Departamentul de Inginerie Biomedică, Școala de Științe ale Vieții, Institutul Național de Știință și Tehnologie Ulsan (UNIST), Ulsan, 44919 Republica Coreea
17 Departamentul de informatică biomedicală, Harvard Medical School, Boston, MA 02115 SUA
Jeremy S. Edwards
18 Chimie și biologie chimică, UNM Comprehensive Cancer Center, Universitatea din New Mexico, Albuquerque, NM 87131 SUA
Jessica A. Weber
19 Departamentul de Biologie, Universitatea din New Mexico, Albuquerque, NM 87131 SUA
Jo Cook
20 Zoological Society of London, Londra, NW1 4RY UK
Sangsoo Kim
21 Departamentul de Bioinformatică și Științele Vieții, Universitatea Soongsil, Seul, 06978 Republica Coreea
Hang Lee
22 Conservation Genome Resource Bank for Korean Wildlife, College of Veterinary Medicine, Seoul National University, Seoul, 08826 Republica Coreea
Andrea Manica
23 Departamentul de Zoologie, Universitatea din Cambridge, Downing Street, Cambridge, CB2 3EJ UK
Ilbeum Lee
24 Daejeon O-World, Daejeon, 35073 Republica Coreea
Stephen J. O'Brien
14 Theodosius Dobzhansky Center for Genome Bioinformatics, St. Universitatea de Stat St. Petersburg, St. Petersburg, 199004 Rusia
25 Oceanographic Center 8000 N. Ocean Drive, Nova Southeastern University, Ft Lauderdale, FL 33004 SUA
Jong Bhak
2 Institutul de Genomică, Institutul Național de Știință și Tehnologie Ulsan (UNIST), Ulsan, 44919 Republica Coreea
3 Departamentul de Inginerie Biomedică, Școala de Științe ale Vieții, Institutul Național de Știință și Tehnologie Ulsan (UNIST), Ulsan, 44919 Republica Coreea
4 Personal Genomics Institute, Genome Research Foundation, Cheongju, 28160 Republica Coreea
5 Geromics, Ulsan, 44919 Republica Coreea
Joo-Hong Yeo
1 Divizia de evaluare a resurselor biologice și genetice, Institutul Național de Resurse Biologice, Incheon, 22689 Republica Coreea
Date asociate
Proiectul de pușcă cu genom întreg leopard a fost depus la DDBJ/EMBL/GenBank sub aderare> LQGZ00000000. Versiunea descrisă în această lucrare este versiunea> LQGZ01000000. Citirile secvențiale ADN brute au fost trimise la baza de date NCBI Sequence Read Archive (SRA321193). Toate datele utilizate în acest studiu sunt disponibile și de pe ftp://biodisk.org/Distribute/Leopard/.
Abstract
fundal
Există trei grupuri dietetice principale la mamifere: carnivore, omnivore și erbivore. În prezent, există o perspectivă comparativă limitată a genomicii asupra evoluției specializărilor dietetice la mamifere. Datorită progreselor recente în tehnologiile de secvențiere, am reușit să efectuăm analize aprofundate ale genomului întreg al reprezentanților acestor trei grupuri dietetice.
Rezultate
Am investigat evoluția carnivorului comparând 18 genomi reprezentativi din toată Mammalia cu specializările dietetice carnivore, omnivore și erbivore, concentrându-ne pe genele Felidae (pisică domestică, tigru, leu, ghepard și leopard), genomidele și genovele Bovidae. Am generat un nou ansamblu de genom de leopard de înaltă calitate, precum și două genomi întregi de leopard sălbatic Amur. În plus față de o contracție clară în familiile genetice pentru metabolismul amidonului și zaharozei, genomurile carnivorelor au prezentat dovezi ale adaptărilor evolutive comune în genele asociate cu dieta, forța musculară, agilitatea și alte trăsături responsabile pentru vânatul cu succes și consumul de carne. În plus, o analiză a regiunilor foarte conservate la nivel de familie a relevat semnături moleculare ale adaptării dietetice în fiecare dintre Felidae, Hominidae și Bovidae. Cu toate acestea, spre deosebire de carnivore, omnivorele și erbivorele au prezentat mai puține semnături de adaptare comune, indicând faptul că carnivorele se află sub o presiune selectivă puternică legată de dietă. În cele din urmă, felidele au prezentat reduceri recente ale diversității genetice asociate cu reducerea dimensiunilor populației, care se pot datora naturii inflexibile a dietei lor stricte, subliniind vulnerabilitatea și starea lor critică de conservare.
Concluzii
Studiul nostru oferă o analiză genomică comparativă la nivel familial la scară largă pentru a aborda modificările genomice asociate specializării dietetice. Analizele noastre genomice oferă, de asemenea, resurse utile pentru cercetarea genetică și de sănătate legată de dietă.
Material suplimentar electronic
Versiunea online a acestui articol (doi: 10.1186/s13059-016-1071-4) conține materiale suplimentare, care sunt disponibile utilizatorilor autorizați.
fundal
Dieta este, poate, cea mai serioasă forță de selecție din toate speciile de pe Pământ. În special, carnivorul este interesant, deoarece a evoluat în mod repetat într-un număr de clade de mamifere [1, 2]. În evidența fosilelor, specializarea în carnivor este adesea asociată cu perioade de dispariție relativ scurte, o consecință probabilă a dimensiunilor reduse ale populației asociate cu o dietă în vârful piramidei trofice [1, 2]. Într-adevăr, mulți specialiști în carnivore au specii strâns legate, care au o dietă mult mai largă, cum ar fi urșii polari, ursii grizzly (omnivori) și ursii panda (erbivori) la Ursidae [3, 4] și vulpile (omnivore) la Canidae [5], evidențiind frecventa instabilitate evolutivă a acestui stil de viață.
Aici, am investigat adaptările genomice la diete extinzând mai întâi acoperirea genomică a Felidae, producând ansamblul genomului de referință pentru pisici mari de cea mai înaltă calitate pentru leopard (Panthera pardus) și datele genomului întreg pentru pisica leopard (Prionailurus bengalensis). Leopardii sunt cele mai răspândite specii de pisici mari (din Africa până în Extremul Orient rus), prosperând într-o mare varietate de medii [20]. Acest ansamblu de leopard oferă un genom suplimentar non-domesticit pentru pisica mare, care poate fi co-analizat cu cea mai precisă referință a genomului pisicii domestice, rezultând studii fiabile de variație genetică la scară genomică pe Felidae. Aceste noi date ne-au permis să comparăm cinci referințe de pisică (pisică domestică, tigru, ghepard, leu și leopard) și doi genomi re-secvențați (leopard de zăpadă și pisică leopard) la un nivel de acoperire comparabil cu alte grupuri bine studiate, cum ar fi hominizii și artiodactilii. Profitând de această bogăție de date, am efectuat o serie de analize comparative pentru a investiga adaptările moleculare la carnivor.
rezultate si discutii
Secvențierea și asamblarea genomului leopardului
Am construit genomul leopardului de referință dintr-un eșantion de mușchi obținut de la un leopard Amur femelă din Daejeon O-World of Korea (Fișier suplimentar 1: Metode suplimentare pentru detalii de identificare a speciilor folosind analiza genei ADN mitocondrial (ADNmt); S1). ADN-ul extras a fost secvențiat la 310 × adâncime medie de acoperire utilizând platformele Illumina HiSeq (Fișier suplimentar 3: Tabelele S1 și S2). Citirile secvențiate au fost filtrate și apoi corectate prin erori folosind o analiză K-mer. S-a estimat că mărimea genomului leopardului este
2,45 Gb (Fișier suplimentar 1: Metode suplimentare pentru detalii; Fișier suplimentar 2: Figura S2; Fișier suplimentar 3: Tabel S3). Citirile corectate de erori au fost asamblate utilizând software-ul SOAPdenovo2 [21] în 265.373 contigs (lungimea N50 de 21,0 kb) și 50.400 schele (lungimea N50 de 21,7 Mb), totalizând 2,58 Gb în lungime (fișier suplimentar 1: metode suplimentare pentru detalii; fișier 3: Tabelul S4). În plus, 393.866 Illumina TruSeq sintetice citesc lung [22] [TSLRs, 2,0 Gb din baze totale;
Analiza evolutivă a carnivorelor în comparație cu omnivorele și erbivorele
Pentru a investiga adaptările genomice la diferite diete și stilurile lor de viață asociate, am efectuat o comparație extinsă de gene ortologice între opt carnivore (leopard, pisică, tigru, ghepard, leu, urs polar, balenă ucigașă și diavolul Tasmanian), cinci omnivore (uman, șoarece, câine, porc și opossum) și cinci genomi erbivori de mamifere (panda uriaș, vacă, cal, iepure și elefant; Fișă suplimentară 1: Metode suplimentare pentru detalii privind criteriile de selecție a speciilor; Fișă suplimentară 3: Tabelul S13). Aceste comparații au dezvăluit numeroase semnături genetice în concordanță cu adaptările moleculare la un stil de viață hipercarnivor.
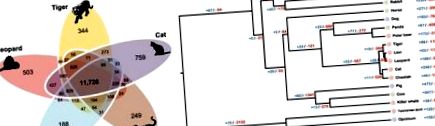
Relația felidelor cu alte specii de mamifere. A Clustere genetice ortologe la speciile Felidae. Clusterele genetice ortologe au fost construite folosind 18 genomi de mamifere. În această figură sunt afișate numai grupurile de gene ale speciilor Felidae. b Expansiunea sau contracția genelor la speciile de mamifere. Numerele de ramură indică numărul de familii de gene care s-au extins (albastru) și s-au contractat (roșu) după despărțirea de strămoșul comun. Culorile cercurilor reprezintă grupuri de diete (roșu deschis: carnivor, albastru deschis: omnivor, verde deschis: ierbivor). Liniile de timp indică timpi de divergență între specii
Evoluția copiei genei și modificările aminoacizilor (AAC) la felide și carnivore. A Familiile contractate (UGT1 și UGT2) și extinse (UGT3) UDP-glucuronosiltransferază din carnivore. Nodurile roșii, violete, albastre și negre sunt gene ale familiei UGT la cele cinci pisici, carnivore care nu sunt pisici (urs polar, balenă ucigașă și diavolul tasmanian), cinci erbivore și, respectiv, cinci omnivori. b AAC convergent găsit în carnivore. Gena embiginei umane (EMB) și structurile proteice prezise sunt ilustrate în partea superioară. Aminoacizii specifici carnivorilor (269 reziduu în proteina EMB umană, regiunea transmembranară) și felidele (309 reziduu, regiune citoplasmatică) din proteina EMB sunt prezentați în roșu și, respectiv, galben. Numerele dintre paranteze reprezintă numărul de genomi analizați în acest studiu
Se știe că pisicilor le lipsește capacitatea de a sintetiza cantități suficiente de vitamina A și acid arahidonic, ceea ce le face esențiale [30]. Interesant este faptul că genele familiei citocromului P450 (CYP), care sunt implicate în catabolismul retinol/acid linoleic/acid arahidonic, au fost contractate în mod obișnuit în toate grupurile dietetice carnivore (Felidae, ordinul Carnivorelor, balena ucigașă și diavolul Tasmanian; Fișa suplimentară 3: Tabelele S18 - S29). Acidul retinoic convertit din retinol este esențial pentru remineralizarea dinților și creșterea oaselor [31, 32], iar acidul arahidonic promovează repararea și creșterea țesutului muscular scheletic după exerciții fizice [33]. Speculăm că contracția genelor familiei CYP poate ajuta carnivorele să păstreze niveluri suficiente de concentrație de retinol și acid arahidonic pe corpul lor și, prin urmare, ar fi putut evolua pentru a poseda mușchi puternici, os și dinți pentru o vânătoare reușită.
De asemenea, am identificat AAC convergente în carnivore (Felidae, urs polar, balenă ucigașă și diavolul tasmanian) și erbivore (panda uriaș, vacă, cal, iepure și elefant). O singură genă embigin (EMB) avea un AAC convergent în carnivore (cu excepția diavolului tasmanian) și nu exista un AAC convergent în erbivore (Fig. 2b), congruent cu sugestia că convergența moleculară adaptivă legată de convergența fenotipică este rară [49 ]. Interesant este că EMB, despre care s-a prezis că va fi modificat funcțional în cele trei clade de carnivore, este cunoscut că joacă un rol în excrescența neuronilor motori și în formarea joncțiunilor neuromusculare [50]. Am confirmat că gena AAC din EMB este conservată și în dihorul intern. În plus, s-a prezis că 18 și 56 de gene sunt funcții specifice carnivorului și respectiv erbivore, modificate de cel puțin un AAC (fișier suplimentar 4: Fișele tehnice S7 și S8). Printre genele modificate de funcția specifică carnivorului, se știe că mai multe gene sunt asociate cu contracția musculară (TMOD4 și SYNC) și sinteza hormonilor steroizi (STAR).
Regiuni foarte bine conservate la nivelul întregii familii
Conservarea secvențelor ADN între specii reflectă constrângeri funcționale și, prin urmare, caracterizarea tiparelor de variație genetică este esențială pentru înțelegerea dinamicii schimbării genomice și adaptarea relevantă a fiecăruia și a unui grup de specii [51, 52]. Am scanat regiuni genomice homozigote, care sunt puternic conservate în rândul speciilor din familii: Felidae (pisică, tigru, leu, ghepard, leopard, leopard de zăpadă și pisică leopard, timp de divergență:
Acum 15,9 milioane de ani [MYA], carnivore), Hominidae (om, cimpanzeu, bonobo, gorilă și orangutan),
15.8 MYA, omnivori) și Bovidae (vacă, capră, oaie, bivoliță de apă și iac,
26 MYA, erbivore) [53-55]. Aceste regiuni extrem de conservate (HCR) reprezintă reducerea variației genetice (regiuni homozigote împărțite între speciile aparținând aceleiași familii; Fig. 3 și fișierul suplimentar 3: Tabelele S39 și S42). Un total de 1,13 Gb de Felidae, 0,93 Gb de Hominidae și 0,88 Gb de Bovidae HCR au fost detectate cu variație genetică semnificativ redusă (P 3 ajustat și Fișierul suplimentar 3: Tabelele S44 și S45) așa cum era de așteptat. Am investigat apoi genele specifice familiei (1436 la Felidae, 2477 la Hominidae și 1561 la Bovidae) în HCR. Genele specifice Felidae s-au îmbogățit semnificativ în percepția senzorială a stimulului luminii (GO: 0050953, 27 gene, P = 0,0022), transmisie sinaptică (GO: 0007268, 33 gene, P = 0,0044), transmiterea impulsului nervos (GO: 0019226, 37 de gene, P = 0,0054) și calea de ghidare a axonului (20 de gene, P = 0,0054; Fișier suplimentar 3: Tabelele S46 și S47), sugerând adaptarea pentru reflexele rapide găsite la pisici. În special, genele specifice Felidae au fost, de asemenea, îmbogățite funcțional pentru procesul biosintetic al carbohidraților (GO: 0016051, 18 gene, P = 0,00061). Acest lucru poate fi legat de tiparul de hrănire prădător al felidelor (o dietă pe bază de carne, deci o disponibilitate dietetică scăzută de carbohidrați). Pe de altă parte, genele specifice Bovidae au fost îmbogățite în percepția senzorială a mirosului (GO: 0007608, 82 gene, P = 2,44 × 10-16) și cognitivă (GO: 0050890, 113 gene, P = 2,54 × 10-9 Fișier suplimentar 3: funcțiile Tabelelor S48 - S50), care indică adaptarea erbivorelor pentru ca mecanismele de apărare să nu fie otrăvite de plante toxice [56].
- DK - Dieta omnivoră - 15 kg Kiezebrink UK Ltd.
- Densitate, destinații sau ambele O comparație a măsurilor de mers pe jos în raport cu transportul
- Efectele unei diete cu conținut scăzut de grăsimi și conținut ridicat de carbohidrați asupra asamblării, producției și trigliceridelor VLDL
- Comparația fitazei microbiene și fosfatului dicalcic pentru creșterea și mineralizarea osoasă a
- Comparație între matrice de diode conectate în linie și detectoare electrochimice în performanțe ridicate