Date asociate
Datele ARN-seq, datele microRNA-seq, datele CNV și datele de metilare a genelor provin din Atlasul genomului cancerului (TCGA). Codurile sursă ale LAceModule sunt disponibile la https://github.com/GaoLabXDU/LAceModule.
Abstract
Introducere
Spre deosebire de corelațiile convenționale (de exemplu, PCC), corelația dinamică se concentrează pe schimbarea corelației a două variabile în urma modificărilor unei a treia variabile (Gunderson și Ho, 2014; Yu, 2018). De exemplu, LA este definit ca media derivatei corelației dintre două obiecte în raport cu o a treia condiție (Li, 2002). LA a fost utilizat pentru a identifica genele candidate la boală (Li și colab., 2007) și genele asociate vârstei umane (Yang și colab., 2018), precum și pentru a descoperi speciile cheie microbiene și factorii de mediu ai comunității microbiene (Ai și colab. ., 2019).
LA este o măsură adecvată pentru evaluarea sensibilității de corelație a ARNc la microARN. În acest studiu, am analizat în primul rând eficacitatea LA în detectarea perechilor de ARNc. Ulterior, am propus un cadru pentru investigarea modulelor ceRNA bazate pe LA (LAceModule) prin integrarea PCC convențională și a corelației dinamice LA cu factor de matrice non-negativă multi-vizualizare (NMF). Prin efectuarea unor analize suplimentare în cancerul de sân, am dezvăluit că ceRNA joacă roluri în aderența celulară, migrarea celulelor și comunicarea celulă-celulă. Rezultatele noastre au arătat, de asemenea, că ceRNA-urile pot reprezenta ținte promitatoare de medicamente și markeri pentru tratamentul și prognosticul cancerului.
Rezultate
LA pentru predicția perechilor ceRNA
Studiile actuale folosesc adesea PCC sau SI pentru a detecta perechi de ARNc. Această abordare ignoră sensibilitatea corelației dintre ARN-uri la nivelurile de expresie ale microARN-urilor lor partajate. Pentru a depăși această limitare, am folosit LA (Li, 2002) pentru a măsura schimbarea dinamică a corelației pentru o pereche de ARNc în funcție de nivelurile de expresie ale microARN-urilor lor partajate. Să presupunem că EXPR1 și EXPR2 reprezintă nivelurile de expresie a doi candidați ceRNA R1 și respectiv R2, în timp ce EXPMIC denotă suma nivelurilor de expresie ale tuturor microARN-urilor lor partajate, MIC. Am normalizat EXPR1 și EXPR2 folosind metoda scorului z astfel încât E (EXPR1) = E (EXPR2) = 0, Var (EXPR1) = Var (EXPR2) = 1, unde E (·) și Var (·) reprezintă așteptarea și varianța unei variabile aleatorii, respectiv.
Presupunând cele de mai sus, PCC între R1 și R2 este:
LA de R1 și R2 în ceea ce privește microARN-urile lor partajate este definit ca LA (R 1, R 2 | MIC) = E (g ′ (EXPMIC)), unde g (EXPMIC) = E (EXPR1 × EXPR2 | EXPMIC = expMIC ). Conform Stein Lemma (Stein, 1981), dacă suma nivelurilor de expresie ale tuturor microARN-urilor partajate MIC urmează distribuția normală standard, LA (R1, R2 | MIC) = E (EXPR1 × EXPR2 × EXPMIC), calculul din LA poate fi simplificat așa cum se arată mai jos:
unde N este numărul eșantionului. Am efectuat transformarea datelor pe EXPMIC folosind metoda Van der Waerden pentru a ne asigura că EXPMIC respectă distribuția normală standard. Pentru EXPMIC1, EXPMIC2, ⋯, EXPMICN, am obținut inițial rangurile lor r1, r2, ⋯, rN și ulterior am calculat valoarea transformată după cum urmează:
unde Φ (·) este funcția de distribuție cumulativă a distribuției normale standard.
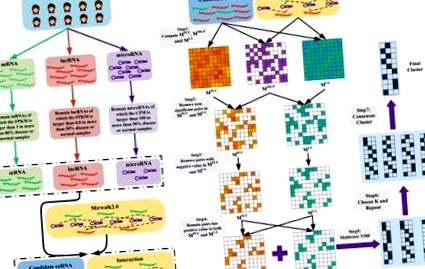
(A) Pregătirea datelor. Am obținut datele ARN-seq ale mARN-urilor și lncRNA-urilor, precum și datele microARN-seq ale microARN-urilor. Ulterior, am îndepărtat ARN-urile neexprimate și slab exprimate. În cele din urmă, am reținut ARN-urile care au fost prezentate în seturile de date de interacțiune ARN-microARN (aici este Mirwalk2.0) ca ceRNA-uri candidate. (B) Prezentare generală a LAceModule. Intrările din LAceModule sunt profiluri candidate de exprimare ceRNA, profiluri de expresie microARN și interacțiuni ARN-microARN. Pentru fiecare pereche de ceRNA candidat, se poate obține valoarea PCC, valoarea LA și gradul de semnificație al microRNA-urilor partajate (MS-P). Pentru perechile cu valori MS-P mai mari (pragul este 0,05), valorile PCC negative sau valorile LA ar trebui eliminate (adică valorile PCC și valorile LA ale acestor perechi sunt setate la zero) . Multi-view NMF este executat folosind matricea PCC, matricea LA și diferite K ca intrări. Cel mai bun K este selectat prin compararea a patru valori de evaluare a grupării. Ulterior, procedurile NMF multi-vizualizare sunt repetate de 10 ori cu cel mai bun K și valori inițiale diferite. Modulele finale sunt obținute prin gruparea consensuală a rezultatelor repetate.
tabelul 1
Valorile LA, PCC și SI ale perechilor ceRNA validate.
| ENSG00000234741 | ENSG00000171862 | −0,058 | 0,040 | −0,008 | 0,005 | BRCA |
| ENSG00000251562 | ENSG00000070831 | 0,043 | −0,009 | 0,002 | 0,001 | BRCA |
| ENSG00000251562 | ENSG00000135446 | −0,377 | 0,000 | −0,003 | 0,022 | BRCA |
| ENSG00000115414 | ENSG00000026508 | 0,082 | −0,003 | −0,001 | 0,001 | BRCA |
| ENSG00000108821 | ENSG00000026508 | −0,014 | 0,082 | 0,001 | 0,029 | BRCA |
| ENSG00000171862 | ENSG00000038427 | 0,379 | 0,075 | −0,004 | 0,002 | BRCA |
| ENSG00000038427 | ENSG00000139687 | 0,368 | 0,058 | 0,000 | 0,003 | BRCA |
| ENSG00000226950 | ENSG00000168036 | 0,131 | 0,103 | −0,003 | 0,012 | LIHC |
| ENSG00000234741 | ENSG00000150593 | 0,205 | −0.205 | −0,014 | 0,003 | LIHC |
| ENSG00000234741 | ENSG00000171862 | −0,003 | −0.107 | −0,002 | 0,013 | LIHC |
| ENSG00000241388 | ENSG00000057663 | 0,035 | −0,068 | −0,005 | 0,033 | LIHC |
| ENSG00000251164 | ENSG00000148516 | −0,093 | 0,097 | −0,001 | 0,004 | LIHC |
| ENSG00000251164 | ENSG00000168615 | −0,392 | 0,41 | 0,003 | 0,034 | LIHC |
(A) Valoarea AUC pentru prezicerea perechilor ceRNA cu LA, PCC și SI în BRCA și LIHC. (B) Evaluarea cluster a trei metode pe diferite matrice din BRCA. (C) Evaluarea cluster a trei metode pe diferite matrice în LIHC. (D) Comparația entropiei de modificare a genei în module între diferite metode de grupare. (E) Compararea microARN-ului mediu validat al fiecărei perechi în module între diferite metode. (F) Comparația dispersiei genelor neregulate în module între diferite metode. Rândul superior: ignorarea direcției disregulării, rândul inferior: luarea în considerare a direcției disregulării. (* p Figura 1B), un cadru bazat pe multi-vizualizare NMF (Liu și colab., 2013) pentru identificarea sistematică a modulelor ceRNA folosind LA. Pentru fiecare pereche de ceRNA candidat, am calculat valoarea PCC, valoarea LA și gradul de semnificație al microARN-urilor partajate (MS-P) (a se vedea secțiunea Materiale și metode) pentru a construi trei matrice M PCC, M LA și M MS - P, respectiv. Ulterior, când valorile MS-P ale perechilor ceRNA candidate au fost ≥0,05, le-am setat valorile corespunzătoare PCC și valorile LA la zero. Datorită cerinței de non-negativitate în cadrul NMF cu mai multe vizualizări, am stabilit valori negative în M PCC și M LA la zero. Având în vedere că o pereche de ARNc ar trebui să fie co-exprimată și sensibilă la schimbarea expresiei microARN-urilor lor partajate, am stabilit valorile din aceeași intrare a M PCC și M LA a perechilor de ARNc candidate la zero dacă oricare dintre aceste valori A fost zero. În cele din urmă, am integrat M PCC și M LA folosind NMF multi-vizualizare pentru a identifica modulele ceRNA.
Pentru NMF multi-vizualizare, există două vederi de observație M = M PCC, M LA>, fiecare dintre ele fiind o matrice G × G non-negativă, unde Gis este numărul de ceRNA-uri candidate. Fiecare matrice din M, M v ∈ M PCC, M LA>, poate fi factorizată la UG × K v ≥ 0 și (VG × K v) T ≥ 0 că M v ≈ U v (V v) T și fiecare rând de (V c) T poate fi considerat ca reprezentarea de rang K a punctului ceRNA candidat corespunzător. Aici, am încercat să identificăm o reprezentare de rang inferior, care este potrivită pentru ambele vizualizări, care este definită ca (V *) T. Am factorizat fiecare matrice în M și am făcut fiecare (V v) T cât mai aproape posibil de (V *) T. Prin urmare, am determinat funcția obiectivă după cum urmează:
unde λPCC și λLA reglează greutatea relativă între diferite puncte de vedere și între eroarea standard NMF și dezacordul dintre (V *) T, (V PCC) T și (V LA) T. Am folosit o procedură iterativă prin actualizarea unei variabile, menținând în același timp variabilele rămase fixate pentru a rezolva această problemă de optimizare (vezi detalii în secțiunea Materiale și metode). După calculul (V *) T, am obținut eticheta modulului ARN i folosind un r g max j = 1, 2, ⋯, K V i j * .
De remarcat, LAceModule necesită predeterminarea numărului de module, K. Am evaluat performanța clusteringului pentru a selecta un K optim variind de la 10 la 400 cu o creștere de 10, luând în considerare patru valori (Figurile 2B, C), și anume C-index (Hubert și Schultz, 1976), McClain-Rao (McClain și Rao, 1975), coeficientul de corelație punct biserială (Milligan, 1981) și coeficientul siluetei (Rousseeuw, 1987). Luând în considerare simultan patru valori pe două matrice, am selectat K = 360 în BRCA și K = 370 în LIHC. Pentru a obține module ceRNA robuste, LAceModule a repetat procedurile multi-vizualizare NMF de 10 ori și a calculat o matrice de consens pentru a identifica modulele ceRNA utilizând algoritmul de partiționare a similarității bazat pe cluster (CSPA) (Strehl și Ghosh, 2003). Mai exact, CSPA generează o matrice binară pentru fiecare rezultat al clusterizării NMF multi-vizualizare, cu „1” reprezentând două gene asociate din același cluster și „0” pentru nu. Matricea consens este suma acestor matrice binare. modulele ceRNA pot fi identificate prin grupare spectrală pe această matrice consens utilizând K optim selectat mai sus.
Comparație între LAceModule și metodele bazate pe PCC/SI
Am folosit NMF pentru a înlocui NMF multi-vizualizare și matricea PCC sau matricea SI ca intrare pentru a compara performanța corelațiilor convenționale și dinamice în detectarea modulelor ceRNA. În matricea PCC și matricea SI, valorile negative sau valorile MS-P corespunzătoare ≥0,05 au fost setate la zero. De asemenea, am testat K variind între 10 și 400, cu o creștere de 10, și am evaluat performanța în cluster cu aceiași indicatori menționați în secțiunea Identificarea modulelor ceRNA folosind LAceModule. Am selectat Ks egale cu 350 și 360 pentru rezultatele bazate pe PCC și SI pe BRCA, în timp ce Ks egale cu 360 și respectiv 340, au fost selectate pentru LIHC (Figurile 2B, C). În secțiunile următoare, am folosit „PCC + LA” pentru a reprezenta modulele detectate de LAceModule, precum și „PCC” și „SI” pentru a reprezenta modulele bazate pe PCC sau respectiv SI, respectiv.
CeRNA-urile sunt reglementate prin microARN-uri partajate. Prin urmare, modulele ceRNA pot avea tendința de a partaja mai mulți microARN-uri în fiecare pereche. Am utilizat interacțiunea ARNm-microARN validată experimental în miRTarBase (Chou și colab., 2016) pentru a evalua numărul mediu de microARN partajate într-o pereche. Rezultatele sunt prezentate în Figura 2E. Modulele „PCC + LA” au împărțit mai multe microARN-uri în medie decât cele din „PCC” (FDR = 1.84E-02 în BRCA, FDR = 1.84E-02 în LIHC; Wilcoxon test cu o singură coadă) și „SI” = 1.05 E-06 în BRCA, FDR = 2.62E-09 în LIHC; test cu o singură coadă Wilcoxon). Mai mult, modulele „PCC” au împărțit mai multe microARN-uri în medie decât cele din „SI” (FDR = 8.46E-03 în BRCA, FDR = 3.82E-05 în LIHC; test cu o singură coadă Wilcoxon).
Colectiv, comparațiile dintre modificarea genelor, raportul de neregulare a genelor și numărul de microARN partajate sugerează că integrarea corelațiilor convenționale și dinamice oferă o detecție mai bună a modulelor ceRNA decât corelația convențională singură.
Analiza funcțională a modulelor ceRNA în cancerul de sân
- Identificarea și cuantificarea componentelor aromelor caracterului în brânza de capră în stil Chevre proaspăt
- Identificarea unei amprente cerebrale pentru supraponderalitate și obezitate - ScienceDirect
- Identificare și profiluri de semnătură pentru mediatori lipidici pro-rezolvatori și inflamatori la om
- Identificarea obezității la pacienții cu diabet de tip 2 din diabetul australian de îngrijire primară
- Fapte despre acarieni; Identificare, control; Prevenirea