Samantha Laber
1 unitate de genetică a mamiferelor, Institutul de cercetări medicale Harwell Institute, Oxfordshire, Marea Britanie
2 Departamentul de Fiziologie, Anatomie și Genetică, Universitatea din Oxford, Oxford, Marea Britanie
Roger D. Cox
1 unitate de genetică a mamiferelor, Institutul de cercetări medicale Harwell Institute, Oxfordshire, Marea Britanie
În ultimii ani, studiile de asociere la nivelul genomului (GWAS) au identificat sute de loci și mii de polimorfisme monoc nucleotidice (SNP) asociate cu diabetul zaharat de tip 2 (T2DM) și trăsături ale obezității [cum ar fi indicele de masă corporală (IMC) și talia –Raportul șoldului (WHR)] în populația umană (1-4). Marea majoritate a acestor SNP se află în regiuni necodificatoare ale genomului și distală de promotori, sugerând că acționează prin reglarea genelor, ceea ce face ca interpretarea lor funcțională să fie dificilă (5). Colectiv, comparând peisajul epigenetic dintre șoareci și om a stabilit noi căi implicate în obezitate și diabet și, de fapt, conservarea inter-specii a fost folosită cu succes ca criterii în găsirea elementelor funcționale și relevante pentru boli (6-8). Prin contrast, analiza comparativă la nivelul genomului a epigenomului de șoarece și uman în țesuturi a evidențiat prezența divergenței cis-reglatoare (9, 10). Noile abordări de inginerie a șoarecilor împreună cu disecția bioinformatică a regiunilor asociate trăsăturilor, de exemplu, modificările epigenetice și interacțiunile genomului au o promisiune mare de a înțelege pe deplin mecanismele de bază ale variantelor necodificate asociate bolii umane în T2DM și obezitate.
Natura specifică contextului semnalelor GWA umane la om
Conservarea între om și șoarece
Conservarea epigenomică între om și șoarece - Insights from the Mouse Encode Consortium
Luate împreună, deși peisajul cis-regulator a divergut substanțial între om și șoarece la nivel global, SNP-urile asociate trăsăturilor umane sunt îmbogățite în situri care sunt conservate între cele două specii pentru majoritatea trăsăturilor investigate.
Potențialul pentru noi strategii în ingineria modelului de șoarece
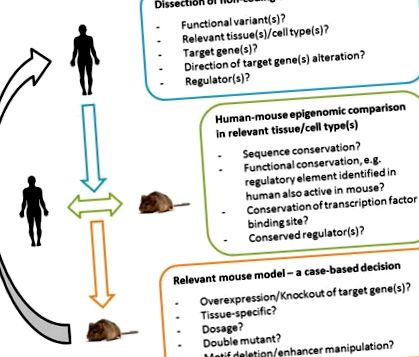
Validarea funcțională a variantelor la om și șoarece. O abordare integrativă pentru generarea de modele de șoarece semnificative și informative ale semnalelor umane ale studiului de asociere a genomului (GWAS). Descifrarea bazelor unui semnal de asociere în contextul uman este esențială. Pentru studii mecaniciste in vivo, o comparație epigenomică de la om la șoarece poate ghida alegerea unui model relevant de șoarece, de exemplu, în cazul conservării funcționale scăzute sau insuficiente a unui sit de reglementare (sau a lipsei seturilor de date care pot determina din urmă) o manipulare clasică a genei țintă specifice țesuturilor se poate dovedi valoroasă; în cazul unei conservări funcționale ridicate (bazată pe disecția genomică și bioinformatică a loci), un model care manipulează selectiv regiunea de reglare poate fi în principiu util. Compararea epigenomului om-șoarece poate fi de așteptat să devină din ce în ce mai puternică odată cu îmbunătățirea calității, precum și cu caracterul cuprinzător al seturilor și instrumentelor de date genomice. Utilitatea de traducere va depinde de modelul mouse-ului ales, iar informațiile obținute pot oferi feedback și ajuta la interpretarea semnalelor GWAS umane.
Concluzie și direcții viitoare
Majoritatea variantelor genetice umane asociate cu trăsăturile comune ale bolilor metabolice se află în elemente de reglare distale. Cu cunoștințele noastre actuale despre reglarea genelor și specificul contextului semnalului, este necesar să înțelegem semnalul la om. Identificarea țintelor și a contextului este crucială în proiectarea unui model relevant de mouse. O comparație epigenomică cuprinzătoare de la om la șoarece poate fi informativă despre variantele de risc uman. Deși intrigant, dacă manipularea elementelor de reglementare va deveni un instrument pentru disecarea variantelor de risc ale obezității umane/T2DM la șoarece va depinde de conservarea funcțională a unui semnal dat. Acest lucru este încă de stabilit și oferă o cale interesantă de explorat.
Glosar
ATAC-seq- Test pentru cromatina accesibilă la transpozază, urmată de o secvențiere de mare viteză. Această tehnică permite identificarea cromatinei deschise.
IMC-Indicele de masa corporala. O măsură a greutății corporale care ia în considerare dimensiunea unei persoane și calculată prin împărțirea greutății corporale la înălțimea pătrată.
ChIP-seq—Imunoprecipitarea cromatinei urmată de o secvențiere de mare viteză. Această tehnică permite identificarea fragmentelor de ADN care sunt legate de un anticorp specific.
Cis-reglementare—Secvențe de ADN necodificate în sau în apropierea unei gene necesare expresiei sale spațio-temporale care conțin caracteristic situsuri de legare a factorului de transcripție.
CRE—Cre recombinaza recunoaște secvențele de ADN cunoscute sub numele de situsuri LoxP și atunci când o pereche de situsuri este furnizată în aceeași orientare, acest lucru duce la ștergerea secvenței intermediare. În acest fel, un segment de ADN, cum ar fi un exon cheie (despre care se spune că este redus) poate fi șters, rezultând, de exemplu, o mutație nulă. Acest lucru se poate face in vivo prin editarea genelor pentru a plasa site-urile LoxP în locația necesară și apoi încrucișarea animalelor care transportă această modificare la tulpinile de recombinază Cre, ceea ce are ca rezultat recombinarea. Expresia recombinazei Cre poate fi condusă de un promotor la alegere fie ca transgenă, fie transformată într-un promotor de gene endogen. Astfel, recombinaza poate fi exprimată în țesuturi specifice, după cum este necesar, permițând recombinarea specifică celulelor sau țesuturilor, adică pentru generarea unui knockout condiționat.
Epigenom—O rețea de compuși chimici (de exemplu, metilarea ADN-ului sau modificări ale histonelor) din jurul ADN-ului care modifică genomul fără a altera însăși secvența ADN. Aceste elemente modificatoare joacă un rol în determinarea genelor care sunt active într-o anumită celulă la un moment dat.
eQTL—Locii trăsăturilor cantitative ale expresiei sunt loci genomici care contribuie la variația nivelurilor de expresie ale ARNm. De exemplu, la indivizii dintr-o populație care moștenește alela SNP A, expresia genei Y se dovedește a fi crescută sau scăzută cantitativ în medie comparativ cu celelalte alele SNP moștenite la nivelul populației testate. Aceasta este mai degrabă o trăsătură corelată decât o legătură funcțională directă între un SNP și expresia unei gene. Mai mult, orice SNP particular marchează un haplotip (un grup legat co-moștenit) de SNP-uri și ca atare reprezintă un locus.
GWAS—Estudiu de asociere la nivel larg.
iPSC—Celula stem pluripotentă indusă.
SNP—Polimorfism cu un singur nucleotid.
T2DM—Diabetul zaharat de tip 2.
Transcriptom—Întregul ARNm exprimat din genele unei celule.
Trans-reglementare—În contextul reglării transcripționale, un element care acționează trans este de obicei o secvență ADN care conține o genă. Această genă codifică o proteină (sau alte molecule precum microARN) care va regla o altă genă țintă.
WHR—Raportul talie-șold.
Contribuțiile autorului
SL și RC au scris, editat și aprobat manuscrisul.
Declarație privind conflictul de interese
Autorii declară că cercetarea a fost efectuată în absența oricărei relații comerciale sau financiare care ar putea fi interpretată ca un potențial conflict de interese.
Mulțumiri
Autorii îi mulțumesc lui Thomas Agnew și Cecilia Lindgren pentru comentariile lor valoroase asupra manuscrisului.
Finanțarea
SL a fost susținut de o bursă de doctorat MRC și cu finanțare RC MRC MC_142661184.
- Genul rezistenței umane, obezitatea și diabetul de tip 2 Diabetul zaharat
- Cum sunt legați îndulcitorii artificiali de diabet și obezitate The Independent The Independent
- Hipogonadismul hipogonadotrop în diabetul de tip 2 și obezitatea
- Cum dansul de sală ajută persoanele cu obezitate și diabet - Fred Astaire Dance Studios Corporate
- La fel ca mazărea și morcovii, modul în care diabetul de tip 2 și obezitatea merg adesea mână în mână