Subiecte
Abstract
Originile staturii impunătoare ale girafei și adaptările cardiovasculare asociate sunt necunoscute. Okapi, care nu are aceste caracteristici unice, este cea mai apropiată rudă a girafei și oferă o comparație utilă, pentru a identifica variația genetică care stă la baza gâtului lung și a sistemului cardiovascular al girafei. Genomurile girafei și okapi au fost secvențiate și, prin analize comparative ale genelor și căilor, au fost identificate care prezintă modificări genetice unice și care probabil contribuie la trăsăturile unice ale girafei. Unele dintre aceste gene se află în căile de semnalizare HOX, NOTCH și FGF, care reglează atât dezvoltarea scheletală, cât și cea cardiovasculară, sugerând că statura girafei și adaptările cardiovasculare au evoluat în paralel prin modificări într-un număr mic de gene. Metabolismul mitocondrial și genele de transport ale acizilor grași volatili sunt, de asemenea, divergenți evolutiv în girafă și pot fi legate de dieta sa neobișnuită, care include plante toxice. În mod neașteptat, s-au produs modificări evolutive substanțiale la girafă și okapi în repararea pauzelor cu două fire și funcțiile centrosomului.
Introducere
Okapi (COM)Okapia johnstoni), ruda cea mai apropiată a girafei și singurul alt membru existent al Giraffidae familie, oferă o comparație utilă, deoarece nu împărtășește aceste atribute unice văzute în girafa 13. Au fost identificate nouă subspecii de girafă care se pot distinge prin culoarea și modelul stratului și au fost izolate reproductiv atâta timp cât 2 mya (ref. 14, 15). Două subspecii de girafe sunt aproape dispărute și, în general, numărul girafelor a scăzut cu 40% din 2000, din cauza braconajului și a pierderii habitatului 16. Deoarece toate subspeciile girafelor împărtășesc adaptarea anatomică și fiziologică unică a genului girafelor, acestea oferă o verificare încrucișată importantă pentru modele unice de variație genetică.
Aici am secvențiat genomul girafei Masai și al okapi și, prin analiza comparativă cu alte mamifere euteriene, au fost identificate 70 de gene care prezintă semne multiple de adaptare (MSA) la girafă. Mai multe dintre aceste gene codifică regulatori binecunoscuți ai dezvoltării scheletice, cardiovasculare și neuronale și sunt susceptibili de a contribui la caracteristicile unice ale girafei.
Rezultate
Secvențierea genomului și de novo asamblare
Analiza comparativă a genomului
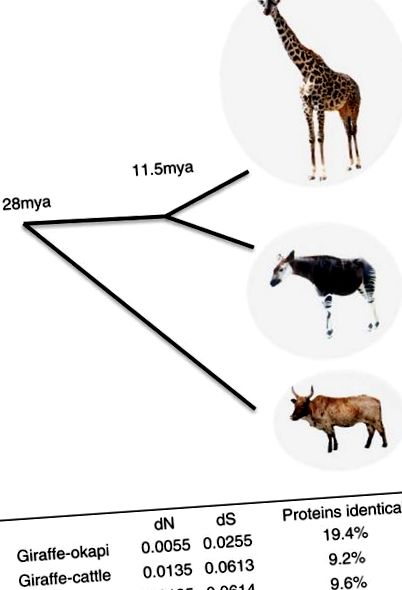
Pentru a identifica modificările care pot sta la baza acestor adaptări morfologice și fiziologice unice, am analizat secvențele de codificare ale genelor ortoloage la girafă, okapi și bovine. Genele girafă și okapi sunt foarte asemănătoare în general, 19,4% dintre proteine fiind identice (Fig. 1). Genele girafei și okapi sunt la fel de îndepărtate de bovine, ceea ce sugerează că caracteristicile unice ale girafei nu se datorează unei rate de evoluție mai rapide. Divergența de girafă și okapi, bazată pe ratele relative de substituții sinonime, de la un strămoș comun este estimată la 11,5 mya (Fig. 1), substanțial mai mică decât estimarea anterioară de 16 mya (ref. 19, 20), care a fost pe baza comparațiilor secvenței ADN mitocondriale.
Utilizând estimările medii de divergență de substituție sinonimă pereche (dS) între girafă, okapi și bovine, calibrate de strămoșul comun pecoran (27,6 mya), divergența girafei și okapi de la un strămoș comun este estimată la 11,5 mya. Imagine Okapi adaptată dintr-o fotografie de Raul654.
Evoluția adaptativă a girafei
Au fost identificate șaptezeci de gene care au prezentat MSA bazate pe divergența secvenței de aminoacizi, evaluată prin analiza filogenetică a vecinilor de proteine ortoloage de mamifere, îmbogățirea substituțiilor nesinonime, substituții unice de aminoacizi la locurile altfel fixate la mamifere, substituții prevăzute pentru a provoca modificări funcționale prin Polifen2 analiză și substituții sub selecție pozitivă. Analiza cluster a fost efectuată pe setul de 70 de gene MSA girafe pe baza procesului biologic GO folosind Cytoscape 3.0 (ref. 68).
Evoluția regulatorilor de creștere și diferențiere a scheletului
Giraffe FOLR1 prezintă dovezi excepțional de puternice pentru evoluția adaptivă, incluzând șase substituții ale aminoacizilor selectate pozitiv, dintre care două se prevede că vor provoca o schimbare semnificativă a funcției (Fig. 3b). FOLR1 mutațiile sunt letale embrionare la șoareci 28 și produc hipomielinizare și defecte neurologice la om 29. În plus față de rolul său în transportul folatului celular, FOLR1 este interiorizat, prelucrat și transportat la nucleu unde reglează componentele căilor FGF și NOTCH 30. Aceste modificări ale girafei FOLR1 pot acționa împreună cu modificări similare în FGFRL1 și JAG1, componentele căilor FGF și, respectiv, NOTCH, pentru a forja adaptări majore de dezvoltare.
Evoluția genelor cardiovasculare și metabolice
Sistemul cardiovascular girafă este adaptat pentru a regla tensiunea arterială la o înălțime de 6 m și pentru a menține homeostazia cardiovasculară asociată cu schimbări rapide în poziția relativă a creierului față de inimă. Tensiunea arterială a girafei este cu 2,5 × mai mare decât cea a omului, ventriculul stâng al inimii este mărit, iar pereții vaselor de sânge ale extremităților inferioare sunt foarte îngroșați 1,31. Girafa prezintă dovezi ale evoluției adaptive a opt gene care reglează tensiunea arterială sau funcția cardiovasculară, inclusiv doi dintre principalii receptori adrenergici α1 și β-2, urotensina-2b și enzima de conversie a angiotensinei (Tabelul suplimentar 4). BORG1 și RCAN3, care sunt extrem de exprimate în inimă și se presupune că au funcții importante legate de forma celulară și, respectiv, contracția mușchilor cardiaci, sunt, de asemenea, semnificativ divergente în girafa 32,33. Modificările distinctive observate în aceste gene pot oferi indicii cu privire la originile evolutive ale tensiunii arteriale crescute ale girafei, creșterii debitului cardiac și vasculaturii modificate.
Statura ridicată a Girafei îi permite să se hrănească cu frunze de salcâm și semințe care sunt foarte hrănitoare, dar conțin și alcaloizi toxici. Ca și în cazul altor rumegătoare, microbii intestinali ai girafelor fermentează plantele pentru a genera acizi grași volatili care sunt transportați prin epiteliul intestinal și servesc drept sursă principală de energie 34.35. Printre genele MSA din girafă sunt incluse cele implicate în catabolismul acizilor grași volatili precum butiratul (MCT1, ACSM3 și ACADS) sau fosforilarea oxidativă din aval care generează ATP (NDUB2 și SDHB) (Fig. 3c). În plus, aceste proteine sunt esențiale pentru transportul și metabolismul lactatului, care este deosebit de important pentru funcțiile cardiovasculare 36 .
Modificări evolutive ale ADN și ale genelor reparatoare ale cromozomilor
Discuţie
Genele reglatoare de dezvoltare și fiziologice la girafă care prezintă evoluție adaptativă sunt îmbogățite în funcții scheletice, cardiovasculare și neuronale. Genele MSA despre care nu se știe că sunt legate de reglarea dezvoltării scheletice, cardiovasculare sau neuronale sunt enumerate (caseta din dreapta).
Dintre cele 70 de gene care prezintă MSA în girafă, FGFRL1 este cel mai puternic candidat pentru a avea un impact direct asupra creșterii unice a scheletului axial și apendicular și a sistemului cardiovascular. FGFRL1 este cunoscut ca fiind esențial pentru dezvoltarea normală a scheletului și cardiovascularului la oameni și șoareci 25,26,27, iar calea FGF reglează dimensiunea somitului 51. Alte gene sunt necesare pentru a restricționa creșterea diferențială la vertebrele și picioarele cervicale, iar genele homeotice, care specifică identitatea diferitelor regiuni ale corpului, joacă probabil acel rol. Am identificat trei gene homeobox -HOXB3, CDX4 și NOTĂ- Care prezintă modificări semnificative la girafă în comparație cu alte mamifere. Apariția metodelor de editare genică oferă un mijloc de testare a acestor ipoteze prin introducerea substituțiilor unice de aminoacizi observate în girafă în genele omoloage ale organismelor model și determinarea consecințelor funcționale. Dintre mamifere, girafa are unele dintre cele mai provocatoare probleme fiziologice și structurale impuse de înălțimea sa falnică. Soluțiile la aceste provocări, în special legate de sistemul său circulator supraalimentat, pot fi instructive pentru tratamentul bolilor cardiovasculare și al hipertensiunii arteriale la om.
Metode
Secvențierea genomului
Kitul de pregătire a bibliotecii fără PCR ADN Illumina TruSeq a fost utilizat pentru a construi biblioteci cu capăt asociat din probe de ficat a două femele de girafă Masai (G.c. tippelskirchi) de la MA1 în Kenya și grădina zoologică din Nashville (NZOO) și un bărbat fetal okapi (O. johnstoni) de la White Oak Holdings. Bibliotecile au fost pregătite în conformitate cu protocolul producătorului folosind 2 μg de intrare și fluxul de lucru cu dimensiunea inserției de 550 bp. Kitul de pregătire a probelor de perechi Nextera Mate a fost utilizat pentru a construi biblioteci de perechi de mate din aceleași trei probe utilizând protocolul „Gel Plus” al producătorului cu selecție de dimensiune de 4–8 kb. Bibliotecile au fost secvențiate pe un Illumina HiSeq 2500 în modul Rapid Run folosind secvențierea de 2 × 150-bp asociată. Toate bibliotecile au fost pregătite și secvențiate de Penn State Genomics Core Facility din University Park, PA. Secvențierea țintită a genelor specifice din Rothschild (G.c. rothschildi) și reticulat (G.c. retulata) girafa a folosit ADN genomic pe care l-am izolat din culturi de celule fibroblastice primare obținute de la Dr. Oliver Ryder de la San Diego Zoo Institute for Conservation Research.
Controlul calității și acoperirea genomului
Variantele de nucleotide interspecifice au fost identificate după cum urmează. Secvențele care s-au aliniat la genomul de referință așa cum s-a descris mai sus au fost sortate după poziția inițială a alinierii lor la genomul de referință. Acestea au fost apoi asamblate folosind o abordare 52 bazată pe referințe, necesitând o acoperire a regiunii de cel puțin 2 ori și cel mult 80 de ori pentru a fi luată în considerare pentru asamblare. Secvențele din probele de okapi au fost aliniate la secvența consensului girafei folosind BWA 53 versiunea 0.5.9 cu argumente implicite și diferențele dintre girafă și okapi au fost apoi identificate folosind SAMtools 54 versiunea 0.1.19 cu argumente implicite și comanda mpileup. Scripturile interne (disponibile la cerere) au fost utilizate pentru a determina poziția variantelor în raport cu secvența de referință (vacă sau câine).
Citirile au fost aruncate dacă procesul de mai sus a evidențiat dovezi ale calității citirii insuficiente sau instabilității regiunii genomice, utilizând trei criterii. În primul rând, citirile au fost necesare pentru a avea o cea mai bună aliniere la ansamblul de referință cu cel puțin 3% mai multe nucleotide identice decât a doua cea mai bună aliniere. În al doilea rând, contigurile de referință au fost ignorate dacă adâncimea de acoperire a fost prea mare sau prea mică în conformitate cu statisticile Lander - Waterman. În al treilea rând, regiunile cu o rată supusă neobișnuit de ridicată a diferențelor între specii au fost ignorate, pentru a diminua impactul duplicărilor și al regiunilor cu complexitate redusă. Adâncimea medie de acoperire a citirii pentru diferențele de nucleotide identificate folosind ansamblul de referință câine și aplicată în analizele ulterioare a fost de 20,0 pentru girafa din MA1, 21,6 pentru girafa din Zoo Nashville (NZOO) și 16,8 pentru okapi.
Aproximativ 300 de gene care au prezentat rapoarte relativ ridicate dN/dS la girafă în comparație cu vaca și okapi nu aveau o acoperire completă față de bovine sau alți ortologi ai altor mamifere. În cele mai multe cazuri, acoperirea incompletă a acestor gene s-a datorat faptului că modelul de referință al genei bovine utilizat a fost incomplet în raport cu alte mamifere. Pentru a completa adnotarea acestor gene, au fost identificate schelele girafei și okapi care conțin aceste gene. Schelele adecvate au fost analizate prin programul de adnotare Gen Same 55 folosind secvențe complete de codificare de referință de la bovine sau umane. Au fost utilizate transcrieri de referință ale ansamblului cu cel mai înalt grad de încredere și informații (TSL: 1, GENECODE basic, APPRIS P1).
Noua asamblare
În primul rând, adaptoarele TruSeq din datele de perechi mate au fost eliminate cu ajutorul parametrilor impliciți Nesoni (v0.115) (https://github.com/Victorian-Bioinformatics-Consortium/nesoni). Apoi, KmerGenie (v1.6269) 56 a fost executat cu parametrii impliciți pe ambele seturi de date, pentru a determina cele mai bune dimensiuni k-mer pentru asamblare. Schelele au fost asamblate folosind SOAPdenovo2 (v2.04) 57, setând dimensiunea k-mer la 91 pentru setul de date girafă și 81 pentru setul de date okapi și activând rezoluția de repetare (parametru -R). În cele din urmă, golurile din schele au fost umplute folosind GapCloser (v1.12) cu parametrii impliciți.
Aceleași citiri de perechi și perechi care au fost folosite pentru asamblare au fost mapate înapoi la ansamblurile girafă și okapi. Programul BWA-MEM a fost executat cu parametrii impliciți și statisticile au fost extrase folosind instrumentul „samtools stats”. Este de remarcat faptul că procentul de cartografiere corespunzătoare a perechilor de mate a fost mai mic decât pentru capetele împerecheate, întrucât intervalul mai mare al unei perechi de mate face mai probabil să se hărțuiască între diferite schele.
Alinieri și arbori genetici
Înainte de alinierea secvențelor, tblastn a fost rulat pe fiecare secvență împotriva secvenței corespunzătoare RefSeq a proteinei de vacă (descărcată de la Ensembl). Acest lucru a asigurat corectarea indel-urilor de schimbare a cadrelor, deoarece s-a observat că unele secvențe au fost de calitate de schiță și pot avea unele erori de secvențiere. Secvențele au fost aliniate utilizând versiunea MUSCLE 3.8 (ref. 58) și arborii filogenetici au fost construiți folosind PhyML Versiunea 3.0 (ref. 59). PhyML folosește un algoritm de căutare a copacilor bazat pe probabilitate pentru a găsi o filogenie optimă. Bootstrapping (n= 100) a fost utilizat pentru a testa robustețea filogeniilor rezultate.
Analize de selecție pozitivă
Evaluarea substituțiilor de nucleotide și aminoacizi
Informatii suplimentare
Coduri de aderare: Date de secvență pentru G. camelopardalis tippelskirchi (MA1 și NZOO) și O. johnstoni (WOAK) au fost depuse în Arhiva de citire scurtă sub numărul proiectului SRP071593 (BioProject PRJNA313910) și codurile de acces NZOO: SRX1624609 și MA1: SRX1624612. Proiectul Whole Genome Shotgun al G. camelopardalis tippelskirchi (MA1) a fost depus la DDBJ/ENA/GenBank sub aderarea LVKQ00000000 și versiunea descrisă în această lucrare este versiunea LVCL01000000. Proiectul Whole Genome Shotgun al O. johnstoni (WOAK) a fost depus la DDBJ/ENA/GenBank sub aderarea LVCL00000000 și versiunea descrisă în această lucrare este versiunea LVCL01000000.
Cum se citează acest articol: Agaba, M. și colab. Secvența genomului girafei dezvăluie indicii privind morfologia și fiziologia sa unice. Nat. Comun. 7: 11519 doi: 10.1038/ncomms11519 (2016).
- Fișă informativă despre girafă Blog Natura PBS
- EXCLUSIVA „A fi Mary Jane” Star Raven Goodwin UREȘTE Eticheta Curvy Girl, dezvăluie noua ei sănătoasă
- Exercițiu pentru pierderea în greutate - Healthsmart Exercise Physiology
- Cheryl Cole își dezvăluie sfaturile despre exerciții și dietă
- Dom Sibley dezvăluie pierderea în greutate de aproape două pietre datorită senzației; conștient de sine; Alunița sportivă