Arlette Pacheco-Sandoval
1 Centrul pentru Investiții și Educație Superior din Ensenada, Ensenada, Baja California, Mexic
Yolanda Schramm
2 Universitatea Autonomă Baja California, Ensenada, Baja California, Mexic
Gisela Heckel
1 Centrul pentru Investiții și Educație Superior din Ensenada, Ensenada, Baja California, Mexic
Elizabeth Brassea-Pérez
1 Centrul pentru Investiții și Educație Superior din Ensenada, Ensenada, Baja California, Mexic
Marcel Martínez-Porchas
3 Centrul pentru investiții în alimente și alimente, Hermosillo, Sonora, Mexic
Asunción Lago-Lestón
1 Centrul pentru Investiții și Educație Superior din Ensenada, Ensenada, Baja California, Mexic
Date asociate
Toate fișierele de secvențiere sunt disponibile din baza de date SRA a NCBI (numerele de acces PRJNA515402 și PRJNA518147).
Abstract
Introducere
Focile de port (Phoca vitulina) sunt pinnipedele cu cea mai largă distribuție din lume, inclusiv oceanele Pacificului de Nord și Atlanticului [1]. Limita sudică a distribuției focii portului din nordul Pacificului (P. v. Richardii) este situată în Mexic pe nouă insule de pe coasta de vest a Baja California: Coronado; Todos Santos; San Martin; San Jeronimo; San Benito; Cedri; Nativitate; Asuncion; și, San Roque. Această limită cuprinde, de asemenea, unele zone de-a lungul litoralului Baja California [2]. Toate insulele sunt relativ aproape de coasta continentală (2-50 km), făcând focile vulnerabile la expunerea la diferite perturbări și poluanți generați de activitatea umană.
Focile de port sunt considerate prădători oportuniste cu un nivel trofic ridicat care se hrănesc cu specii bentonice sau pelagice sau ambele specii predominant pești și cefalopode [3]. Focile portului mexican se hrănesc la adâncimi mici, între 5–50 m [4,5], în principal pe pradă bentonică sau demersală [6-9]. Dintre pinipedele găsite în Mexic, foca portului a permis obținerea celei mai mari cantități de informații cu privire la calitatea mediului lor local, datorită fidelității lor față de siturile de transport, unde rămân tot anul [2]. Pe baza datelor emițătorului prin satelit, se știe acum că focile portului se hrănesc în apropierea coloniilor lor (la o distanță între 25 km [10] și 30 km [4]); prin urmare, sunt de așteptat diferențe în tipul de pradă consumată [8,9], deoarece majoritatea coloniilor de foci din Baja California sunt separate de distanțe mai mari. Cu toate acestea, s-au constatat diferențe în dietă și în coloniile separate de numai 16 km [7]. Această variabilitate a dietei se poate reflecta în microbiota focilor, care poate dezvălui și prezența substanțelor nocive în mediu.
Dieta cu focă de port este bogată în proteine și acizi grași polinesaturați [11], care se așteaptă să se reflecte în compoziția sa microbiotică intestinală [12]. După cum sa stabilit de mult, dieta determină în mare măsură compoziția comunității de microbiote intestinale [13,14] și este considerată principala cale de colonizare microbiană [15]. Cu toate acestea, există prejudecăți bine cunoscute în tehnicile tradiționale de analiză a dietei, în special în identificarea resturilor de pradă, datorate eroziunii digestive [16]. Tehnicile ADN dezvoltate recent, cum ar fi metabarcodarea ADN-ului, au identificat prada care nu lasă rămășițe dure (de exemplu, crustacee), oferind informații mai precise despre intervalele dietetice [16].
Microbiota intestinală joacă un rol esențial în dezvoltarea ontogenetică a gazdei, fiziologia, sănătatea, fitnessul și asimilarea nutrienților [17,18]. În ciuda importanței sale, se știe puțin despre compoziția microbiotei și funcțiile sale la mamiferele marine, în parte din cauza dificultăților de colectare a probelor. Diverse studii au examinat microbiota intestinală la mamifere captive sau sălbatice, cum ar fi dugongii [19], lamantinul [20], balenele [21] și pinipedele [12,22-26], relevând o bogăție mai mare a microorganismelor decât cea găsită în omologii terestri, care s-ar putea datora achiziției taxonilor bacterieni marini [27]. Din câte știm, doar un studiu a explorat microbiota intestinală a focilor de port, dar la indivizii semi-captivi [12]. Având în vedere că compoziția microbiană este puternic influențată de factorii de mediu, cercetările existente, întreprinse asupra animalelor captive, oferă puține posibilități de examinare a efectelor microbiotei intestinale asupra metabolismului gazdei în habitatele naturale ale individului, unde factori, precum disponibilitatea alimentelor și clima, variază [28].
Cercetările efectuate pe primate neumane au arătat că studiile asupra microbiotei intestinale sunt utile ca indicatori ai sănătății gazdei și ar putea ajuta la detectarea diferențelor genetice între populații [29]. Deoarece compoziția microbiană a indivizilor din aceeași populație are mai multe asemănări decât cea a diferitelor populații [29], ne-am așteptat să vedem diferențe între comunitatea de microbiote din coloniile de focă port prezentate aici. Scopul acestui studiu a fost de a caracteriza comunitatea de microbiote intestinale a focilor de port din Baja California, prin secvențierea regiunii V4 a ADN-ului microbian ribozomal 16S prelevat din probe fecale, pentru a înțelege rolul dietei în modelarea microbiomului intestinal. Am evaluat relația dintre microbiomul intestinal și dietă ca o primă abordare pentru a înțelege modul în care prada consumată modelează compoziția și funcția comunității microbiene intestinale în focile sălbatice ale portului. Aceste informații pot poziționa, de asemenea, sigiliul portului ca specie santinelă de mediu pentru coasta Baja California și insulele sale.
Materiale și metode
Declarații de etică
Toate probele au fost colectate fără a deranja vreun animal sau a le provoca stres sau rău. Autorizațiile au fost furnizate de Ministerul Mexicului pentru Mediu și Resurse Naturale (SEMARNAT, SGPA/DGVS/12269/13 și SGPA/DGVS/08370/14), Ministerul de Interne (SEGOB, UG/211/0087/2014 și UG/211/01022/2014) și Rezervația Biosferei El Vizcaino (F00.DRPBCPN.000027 și F.00.1.DRPBCPN.00004/2014).
Colectie de mostre
Douăzeci de eșantioane fecale au fost colectate din mai până în iunie 2014 în cinci colonii de foci situate în Baja California, Mexic: Estuarul Punta Banda (PBE) (n = 5); Insula Todos Santos (TS) (n = 3); Insula San Jeronimo (SJ) (n = 4); Insula Natividad (N) (n = 4); și Insula San Roque (SR) (n = 4) (S1 Fig.). Coordonatele plajelor specifice în care au fost colectate probe sunt disponibile în metadatele proiectului de secvențiere din depozitul bazei de date. Probele au fost colectate în timpul sezonului de năpârlire, când cel mai mare număr de indivizi se îndepărtează [30] având tendința de a rămâne pe mal multe ore [31]. Fiecare probă a fost colectată individual folosind o spatulă sterilizată de unică folosință. Probele au fost obținute din partea interioară a fecalelor într-un efort de a evita contaminarea încrucișată din mediul înconjurător. Probele fecale au provenit din sigilii fără distincție de sex și categorii de vârstă, doar probe proaspete plasate în tuburi sterile și apoi conservate în RNAlater® (Sigma-Aldrich). Probele au fost transportate la laborator și ținute congelate la -80 ° C până la o analiză ulterioară.
Extracția și secvențierea ADN-ului
ADN-ul genomic a fost extras din 20 de probe individuale de fecale, inclusiv (PBE = 5, TS = 3, SJ = 4, N = 4 și SR = 4) folosind QIAamp® DNA Stool Mini Kit (QIAGEN). Din fiecare probă, s-au utilizat aproximativ 300-400 mg de material fecal. Calitatea ADN-ului a fost vizualizată prin electroforeză pe gel de agaroză și concentrația a fost determinată folosind un spectrofotometru Nanodrop® (Thermo Fisher Scientific).
Regiunea hipervariabilă V4 a genei 16S rARN a fost amplificată prin intermediul unei metode în două etape de reacție în lanț a polimerazei (PCR). PCR inițială a fost efectuată cu ADN șablon de 10 ng într-o reacție de 20 μl folosind primeri specifici regiunii: 16S-F (5'- GTGCCAGCMGCCGCGGTAA- 3 ') și 16-R (5'– GGACTACHVGGGTWTCTAAT- 3') [32 ]. Programul PCR a constat într-o denaturare inițială la 95 ° C timp de 3 minute și 25 de cicluri la 95 ° C timp de 30 s, 55 ° C timp de 30 s și 72 ° C timp de 30 s, cu o extensie finală la 72 ° C timp de 5 min. Al doilea PCR a fost efectuat folosind primerii dintr-un kit index Nextera XT (Illumina) cuprinzând opt cicluri (95 ° C x 30 s; 61 ° C x 30 s; 72 ° C x 5 min). Reacțiile PCR au fost efectuate individual pentru fiecare probă, iar produsele au fost vizualizate prin electroforeză pe gel și cuantificate cu un kit Qubit dsDNA BR Assay (Thermo Scientific) pe un fluorometru Qubit 3.0 (Thermo Fisher Scientific). Normalizarea produselor PCR a fost efectuată folosind kitul SequalPrep TM Normalization Plate (96) (Applied Biosystems ™) conform protocolului producătorului pentru a obține fiecare amplicon la o concentrație de 1-2 ng/μl înainte de amestecare.
Rezerva de probe (1100 pM), care conținea 10 μl din fiecare probă, a fost denaturată cu NaOH 0,2N, diluată în continuare până la 5 PM și combinată cu 5% (v/v) PhiX 5 pM denaturată, pregătită respectând ghidurile Illumina . Secvențierea a fost efectuată la instalațiile de secvențiere ale Centrului de Cercetare Științifică și Învățământ Superior de la Ensenada (CICESE) utilizând platforma de secvențiere MiSeq (Illumina). Chimia utilizată a fost MiSeq Reagent Kit v2, producând 2x150 bp citiri cu capăt asociat. Măsurile inițiale de control al calității au inclus îndepărtarea oricărei secvențe care conține un nucleotid nerezolvat și secvențe scurte (−20, o identitate procentuală ≥ 96% și o lungime de aliniere de ≥ 100 bp. Sarcinile de specii obținute au fost inspectate manual pentru a elimina speciile care nu au fost distribuite în zona de studiu, contaminanții identificați, speciile care nu erau o pradă potențială (animale terestre, microorganisme etc.) și secvențe de focă. Bazele de date utilizate pentru a determina distribuția și obiceiurile prăzii identificate au fost FishBase (http: // www .fishbase.org/search.php) și SealLifeBase (http://www.sealifebase.org/). Am folosit pachetul R UpsetR [44] pentru a vizualiza numărul de indivizi pradă, atât partajate, cât și unice printre sigiliul portului. colonii.
Predicția funcțională
Alegerea OTU de referință închisă a fost efectuată în baza de date de referință GreenGenes (versiunea 13.5) în QIIME. Pachetul software bioinformatică al Langille Lab, PICRUSt (investigație filogenetică a comunităților prin reconstrucția stărilor neobservate) [45], a fost aplicat pe serverul Galaxy și a fost utilizat pentru a prezice compoziția metagenomului comunității prin intermediul datelor de secvențiere 16S rDNA. Odată ce numărul de copii 16S a fost normalizat și funcțiile metagenomului au fost prezise, căile KEGG (Kyoto Encyclopedia of Genes and Genomes) au fost utilizate pentru identificarea conținutului metagenomic. Comparații statistice au fost efectuate în software-ul R [36] folosind un test ANOVA urmat de un Tukey HSD pentru test de comparație multiplă.
Relația dintre dietă și microbiota intestinală
Pentru a evalua relația dintre dietă și funcția microbiană intestinală și compoziție, am aplicat teste de corelație Spearman între frecvența apariției prăzii, după cum se indică prin abundența relativă a bacteriilor (cele mai abundente filuri și cele 10 familii detectate cel mai frecvent) și abundența relativă a căilor metabolice. Familiile și speciile de pradă cu o frecvență de apariție> 20% au fost incluse în timp ce p Fig 1A) (tabelul S1). Dominația acestor filuri în microbiota intestinală a focii portului a fost raportată anterior [12,25]. La fel ca alte mamifere marine, foca portului prezintă o abundență mai mică a firului Firmicutes decât cea prezentată de mamiferele terestre [26]. O proporție mai mare de Firmicutes decât Bacteroidetes a fost legată atât de o eficiență mai mare în recoltarea energiei din alimente, cât și de apariția obezității la oameni și șoareci [46]. Raportul ridicat Firmicutes la Bacteroidetes observat în intestinul focilor portului s-ar putea datora faptului că aceste animale se bazează în mare măsură pe depunerea grăsimilor pentru stocarea energiei și termoreglare [22,47]. Astfel, este posibil ca focile port să se bazeze pe microbii intestinali pentru a facilita depozitarea eficientă a grăsimilor necesare pentru supraviețuire, prin disponibilitatea crescută a energiei colonice, așa cum se observă la om [48].
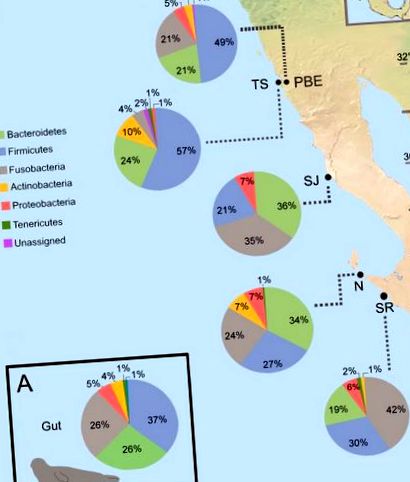
(A) Compoziția generală a microbiotei intestinale la nivelul filelor. (B) Abundența relativă medie a celor mai frecvente filuri bacteriene din coloniile de foci port, sunt arătați doar taxonii cu> 1% abundență relativă. Locațiile de eșantionare a focilor portuare de pe coasta de vest a Baja California sunt incluse în figură ca PBE = Punta Banda Estuary, TS = Todos Santos Island, SJ = San Jeronimo Island, N = Natividad Island, SR = San Roque Island.
La nivel familial bacterian, 19 familii au fost identificate cu o abundență relativă de ≥ 1% și au inclus ordinul bacterian Clostridiales, deși nu a fost clasificat la nivel familial. Fusobacteriaceae a fost cea mai abundentă familie (27%), urmată de Ruminococcaceae (16%) și Bacteroidaceae (14%) (Fig 2). Membrii familiei Ruminococcaceae au prezentat o puternică corelație pozitivă cu trigliceridele polinesaturate de origine dietetică [49], care sunt abundente în prada de focă, cum ar fi hamsia și sardina.
Sunt afișate numai taxonii cu o abundență relativă> 1%. PBE = Estuarul Punta Banda, TS = Insula Todos Santos, SJ = Insula San Jeronimo, N = Insula Natividad, SR = Insula San Roque.
Fusobacterium (filus Fusobacteria), un gen cunoscut pentru manifestarea activității proteolitice în intestinul uman [47], s-a dovedit a fi genul predominant în microbiota intestinală a focilor portuare (25%). Mamiferele marine au o abundență mai mare de Fusobacterii decât mamiferele terestre [26]. Membrii acestui filum, precum Fusobacterium, locuiesc în cavitatea bucală și în intestinul animalelor, precum și se găsesc în sedimentele marine [18]. Focile de port pot dobândi aceste microorganisme din microbiota sedimentului deoarece se hrănesc în principal cu pradă bentonică și demersală (de exemplu, pești plat) [6,50]. Achiziționarea acestor microbi ar putea aduce gene utile noi din microbiomul global în microbiomul intestinal prin transfer orizontal de gene [18].
În cadrul filului Bacteroidetes, genul Bacteroides (14%) și alți reprezentanți ai familiilor Rikenellaceae (7%) și Paraprevotellaceae (6%) s-au dovedit a fi abundente. Genul Bacteroides a fost legat de dietele carnivore, caracterizate prin niveluri ridicate de proteine, aminoacizi și grăsimi animale [51]. Codificând un număr mare de enzime active de carbohidrați derivate din animale, bacterioizii au o capacitate mare de a hidroliza glicanii dietetici, un tip de polizaharide derivate din proteine [52]. Prin urmare, abundența ridicată a genului Bacteroides găsită în focile portului prin prezenta cercetare este în concordanță cu obiceiurile lor carnivore. Enzimele conținute în aceste tipuri de bacterii pot ajuta sigiliul portului să obțină energie din prada consumată [51].
Firmicutes au fost reprezentate de membri ai familiilor Ruminococcaceae (14%), Clostridiaceae (3%), Lachnospiraceae (1%), Mogibacteriaceae (1%) și Peptostreptococcaceae (1%) și de genurile Clostridium (3%), Oscillospira (1%), Faecalibacterium (1%) și Phascolarctobacterium (1%). Familiile Ruminococaceae și Lachnospiraceae joacă roluri importante în funcțiile imune ale gazdei [53] și se găsesc frecvent în tractul digestiv al multor specii de mamifere, în timp ce populațiile abundente sunt absente în mediile neasociate gazdei [54-56].
Filul Proteobacteria a fost reprezentat în principal de genurile Cupriavidus (1%) și Anaerobiospirillum (1%) și de un membru al familiei Vibrionaceae (1%). Într-o măsură mai mică, am găsit membri ai acestui filum care nu sunt considerați în mod obișnuit ca prezenți în tractul digestiv al animalelor de mamifere, genurile Photobacterium (0,004%) și Vibrio (0,0005%), care sunt raportate în abundență mai mare la animalele cu bază de calamar diete, cum ar fi balenele pilot [57]. Bacteriile luminescente Photobacterium și Vibrio sunt membri obișnuiți ai microbiotei de calmar și pot fi dobândite de focile portului prin consumul de calamar [58]. Atopobium (3%) și Slackia (1%) au fost principalii reprezentanți ai filului Actinobacteria.
Comparație între colonii
Deoarece compoziția microbiană intestinală a indivizilor din aceeași populație prezintă mai multe asemănări decât cea a indivizilor din populații diferite [29], ne-am așteptat să găsim diferențe în microbiota diferitelor colonii de foci port studiate aici. Rezultatele noastre au arătat variații între coloniile de foci de port în ceea ce privește abundența relativă a filelor dominante (S2 Fig). Dintre cele 26 de filme observate, cinci au contribuit> 2% la diferențele găsite între colonii, pe baza rezultatelor obținute din analiza SIMPER (Tabelul 1). Modificările abundenței relative a membrilor Firmicutes (33,88%), Fusobacteria (31,95%) și Bacteroidetes (16,86%) explică în principal diferențele de diversitate observate (Tabelul 1). Conform analizei SIMPER, la nivel familial Fusobacteriaceae (25,93%) și Ruminococcaceae (13,19%) au contribuit cel mai mult la diferențele de microbiotă intestinală găsite în coloniile de focă port (Tabelul 1). Membrii familiilor Ruminococcaceae și Lachnospirace sunt frecvente în intestinul mamiferelor și se știe că transformă carbohidrații în acizi grași cu lanț scurt (SCFA) prin fermentație microbiană, cu produsul final absorbit de gazdă ca sursă de energie [59].
tabelul 1
PBE = Estuarul Punta Banda, TS = Insula Todos Santos, SJ = Insula San Jeronimo, N = Insula Natividad, SR = Insula San Roque.
- Relația complexă dintre dietă și sănătate - PubMed
- Relația complexă dintre dietă și sănătate
- Creșterea dietei conduse de nutriționiști Cum mi-am schimbat relația cu alimentele și mi-am atins obiectivul
- De ce pot; t Pierd în greutate în ciuda dietei și a exercițiilor fizice Institutul de medicină funcțională
- The Fiji Times; Ancient Fiji Dieta insulei Pacific