Giulia E Tyzack
1 Institutul Francis Crick, 1 Midland Road, Londra, Marea Britanie
2 Departamentul de boli neuromusculare, UCL Queen Square Institute of Neurology, Queen Square, Londra, Marea Britanie
Raphaelle Luisier
1 Institutul Francis Crick, 1 Midland Road, Londra, Marea Britanie
Doaa M Taha
1 Institutul Francis Crick, 1 Midland Road, Londra, Marea Britanie
2 Departamentul de boli neuromusculare, UCL Queen Square Institute of Neurology, Queen Square, Londra, Marea Britanie
Jacob Neeves
1 Institutul Francis Crick, 1 Midland Road, Londra, Marea Britanie
2 Departamentul de Boli Neuromusculare, UCL Queen Square Institute of Neurology, Queen Square, Londra, Marea Britanie
Miha Modic
1 Institutul Francis Crick, 1 Midland Road, Londra, Marea Britanie
2 Departamentul de boli neuromusculare, UCL Queen Square Institute of Neurology, Queen Square, Londra, Marea Britanie
Jamie S Mitchell
1 Institutul Francis Crick, 1 Midland Road, Londra, Marea Britanie
2 Departamentul de Boli Neuromusculare, UCL Queen Square Institute of Neurology, Queen Square, Londra, Marea Britanie
Ione Meyer
2 Departamentul de boli neuromusculare, UCL Queen Square Institute of Neurology, Queen Square, Londra, Marea Britanie
Linda Greensmith
2 Departamentul de boli neuromusculare, UCL Queen Square Institute of Neurology, Queen Square, Londra, Marea Britanie
Jia Newcombe
3 NeuroResource, Departamentul de Neuroinflamare, UCL Queen Square Institute of Neurology, Queen Square, Londra, Marea Britanie
Jernej Ule
1 Institutul Francis Crick, 1 Midland Road, Londra, Marea Britanie
2 Departamentul de boli neuromusculare, UCL Queen Square Institute of Neurology, Queen Square, Londra, Marea Britanie
Nicholas M Luscombe
1 Institutul Francis Crick, 1 Midland Road, Londra, Marea Britanie
4 UCL Genetics Institute, University College London, Gower Street, Londra, Marea Britanie
5 Institutul Okinawa de Știință și Tehnologie Universitatea Absolventă, Okinawa 904-0495, Japonia
Rickie Patani
1 Institutul Francis Crick, 1 Midland Road, Londra, Marea Britanie
2 Departamentul de boli neuromusculare, UCL Queen Square Institute of Neurology, Queen Square, Londra, Marea Britanie
Date asociate
Datele care susțin rezultatele acestui studiu sunt disponibile de la autorul relevant, la cerere rezonabilă.
Abstract
A se vedea Vidal și Atkin (doi: 10.1093/brain/awz256) pentru un comentariu științific la acest articol.
Introducere
Scleroza laterală amiotrofică (SLA) este o afecțiune neurodegenerativă progresivă neîncetat, care rămâne incurabilă datorită înțelegerii noastre incomplete a patogenezei moleculare. Descoperirile genetice din SLA implică puternic regulatori de prelucrare a ARN-ului exprimate omniprezent (Taylor și colab., 2016). Din punct de vedere patologic, în 97% din cazuri, proteina TDP-43 este localizată greșit de la nucleu la citoplasmă, unde se agregă (Neumann și colab., 2006). Cu toate acestea, mutațiile cauzatoare de SLA în fuziunea în sarcom (FUS) și superoxid dismutaza 1 (SOD1) lipsesc în mod vizibil proteinopatie TDP-43 în majoritatea cazurilor (Mackenzie și colab., 2007). Agregarea FUS este o caracteristică recunoscută a SLA legată de mutația FUS (Vance și colab., 2009). Cu toate acestea, conceptul conform căruia mislocalizarea nucleară către citoplasmatică FUS de tip sălbatic (mai degrabă decât agregarea) ar putea fi o caracteristică mai răspândită a altor forme de SLA nu a fost evaluată în mod sistematic la cunoștința noastră. Într-adevăr, o astfel de localizare greșită poate să fi evitat detectarea până acum datorită tendinței spre studierea agregatelor/incluziunilor, mai degrabă decât localizării subcelulare perturbate a proteinelor neagregate singure. Această posibilitate este întărită de faptele care (i) afectează compartimentarea nuclearo-citoplasmatică fiind din ce în ce mai recunoscută ca o caracteristică cheie a SLA (Boeynaems et al., 2016); și (ii) FUS este cunoscut pentru a naveta între nucleu și citoplasmă.
Aici am investigat sistematic localizarea proteinei FUS pe un model de celule stem pluripotente induse de om (iPSC), modele transgenice de șoarece și țesut uman post-mortem din mai multe cazuri de SLA sporadică. Găsim că mislocalizarea nucleară-citoplasmatică a FUS este o caracteristică mai răspândită a SLA decât s-a recunoscut anterior. Mai mult, prezentăm dovezi care susțin un mecanism molecular presupus pentru această mislocalizare prin interacțiunea dintre proteina FUS și transcrierea SFPQ aberantă care păstrează intronul.
Materiale și metode
Cultură de celule stem pluripotente induse și diferențierea neuronilor motori
IPSC-urile au fost menținute folosind protocoale standard. Diferențierea neuronilor motori a fost efectuată așa cum s-a descris anterior (Hall și colab., 2017; Luisier și colab., 2018). Consultați Material suplimentar pentru informații mai detaliate. Detaliile liniilor iPSC sunt furnizate în Tabelul 1 suplimentar.
Animale, modele transgenice și prelucrarea țesuturilor
Țesut uman post-mortem
Secțiunile de țesut înghețat rapid au fost obținute de la măduva spinării lombare a opt donatori sănătoși și a pacienților SLA sporadici cu vârsta și sexul de 12 ani (tabelul suplimentar 2). Timpul de întârziere până la înghețarea rapidă a fost, de asemenea, comparabil între grupuri [întârziere medie ± deviație standard (SD): 30,13 ± 12,87 și 27,75 ± 10,63 h, pentru pacienții martor și respectiv SLA sporadici]. Probele măduvei spinării au fost obținute de la banca de țesuturi NeuroResource, UCL Institute of Neurology, Londra, Marea Britanie. Probele au fost donate băncii de țesuturi cu consimțământul scris al donatorului de țesuturi, în urma examinării etice a Comitetului NHS NRES Londra - Central și stocate sub o licență sectorială de cercetare de la Autoritatea pentru țesuturi umane (HTA) din Marea Britanie. În acest studiu, cazurile de SLA au fost considerate sporadice de absența oricărui istoric familial de boală a neuronilor motori. Mai mult, toate cazurile au arătat patologia TDP-43 atunci când au fost examinate de NeuroResource, sugerând că nu sunt mutanți SOD1 [deoarece patologia TDP-43 este absentă în cazurile de SLA legate de SOD1 (Mackenzie și colab., 2007)].
Imunomarcare, imagistică și analiză a imaginii
Fracționarea celulelor, extracția ARN, transcrierea inversă și PCR cantitativă
Hibridizare in situ a fluorescenței cu o singură moleculă
Sondele au fost proiectate folosind software-ul Probe Designer de la Biosearch Technologies și au fost furnizate de același furnizor. Probele incluse au fost concepute împotriva intronului SFPQ conjugat cu Quasar®570 (SMF-2037-1) și SFPQ matur conjugat cu Quasar®670 (secvențe de probe disponibile la cerere). Cuantificarea semnalului de hibridizare a fost efectuată utilizând algoritmul personalizat de detectare a intensității spotului în celulele segmentate DAPI pentru a separa semnalul nuclear și citoplasmatic.
Analiza datelor despre localizarea subcelulară a proteinei FUS
Am folosit R și lme4 (Bates și colab., 2015) pentru a efectua analize de efecte mixte liniare ale relației dintre localizarea FUS și mutația VCP sau SOD1, precum și cu SLA sporadică, care reprezintă variații idiosincratice datorate fie diferențelor animale, fie individuale. . Ca efecte fixe, am introdus mutația sau variabila bolii SLA în model. Ca efecte aleatorii, am avut interceptări fie pentru animale (SOD1, VCP), fie pentru pacienți și loturi. Inspecția vizuală a parcelelor reziduale nu a relevat nicio abatere evidentă de la homoscedasticitate sau normalitate. Valorile P au fost obținute prin teste ale raportului de probabilitate ale modelului complet cu efectul în cauză împotriva modelului fără efectul în cauză.
Cartarea datelor iCLIP
Datele brute de legare încrucișată și imunoprecipitare (iCLIP) cu rezoluție individuală a nucleotidelor FUS pot fi accesate la https://imaps.genialis.com/ (Attig et al., 2018) Înainte de aliniere, îndepărtarea adaptorului în două trepte a fost efectuată folosind Cutadapt conform procedurii de operare standard ENCODE iCLIP. O abordare în două etape a fost, de asemenea, utilizată pentru alinierea. În primul rând, Bowtie2 a fost utilizat pentru a elimina citirile aliniate la ARNr sau ARNt. Apoi, STAR a fost folosit pentru a alinia citirile rămase la GRCh38, cu numai citirile de mapare unice păstrate. Duplicatele PCR au fost prăbușite pe baza identificatorilor moleculari unici și a locațiilor de mapare. Poziția reticulării cu rezoluție nucleotidică a fost calculată ca coordonată imediat precedentă evenimentului de trunchiere a transcripției inverse.
Respectarea standardelor etice
Pentru munca iPSC umană, în acest studiu a fost obținut consimțământul informat de la toți pacienții și controale sănătoase. Protocoalele experimentale au fost efectuate în conformitate cu reglementările și ghidurile aprobate de către Spitalul Național de Neurologie și Neurochirurgie al UCLH și de comitetul mixt de etică al cercetării Institutului Queen Square al UCLH (09/0272). Probele umane de măduvă spinării post-mortem au fost obținute de la banca de țesuturi NeuroResource, UCL Queen Square Institute of Neurology, Londra, Marea Britanie. Probele au fost donate băncii de țesuturi cu consimțământul scris al donatorului de țesuturi, în urma examinării etice a Comitetului NHS NRES Londra - Central și stocate sub o licență sectorială de cercetare de la Autoritatea pentru țesuturi umane (HTA) din Marea Britanie. Toate experimentele pe animale descrise în acest studiu au fost efectuate sub licență de la Home Office din Marea Britanie și au fost aprobate de Comitetul de revizuire etică al Institutului de neurologie.
Disponibilitatea datelor
Datele care susțin constatările acestui studiu sunt disponibile de la autorul relevant, la cerere rezonabilă.
Rezultate
Mislocalizarea nucleară către citoplasmatică a FUS la modelele ALS mutante VCP umane și de șoarece
Mislocalizarea nucleară către citoplasmatică a FUS în SLA sporadică umană
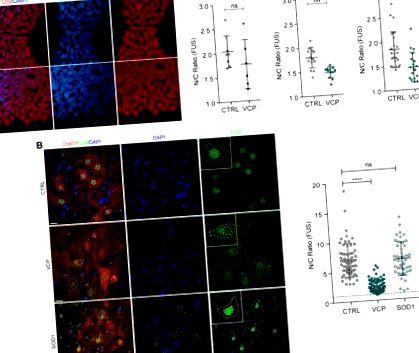
După ce am stabilit că FUS este localizat greșit în modelele ALS legate de VCP, dar nu și în SOD1, am urmărit în continuare să abordăm generalizabilitatea acestei descoperiri între formele sporadice de SLA (care reprezintă 90% din toate cazurile). În acest scop, am examinat țesutul măduvei spinării post-mortem din 12 cazuri SLA sporadice și opt controale sănătoase (Fig. 2 A). Am găsit dovezi clare ale mislocalizării FUS nuclear-citoplasmatic în aceste cazuri SLA sporadice, dar în absența incluziunilor FUS citoplasmatice.
Mislocalizarea nucleară către citoplasmatică a FUS în SLA sporadică umană. (A) Analiza localizării subcelulare a FUS în neuronii motori din măduva spinării ventrală a controalelor sănătoase (n = 8) și a pacienților cu SLA sporadică (sALS) (n = 12). Citoplasma neuronului motor a fost identificată prin imunomarcare ChAT, nucleele au fost contracolorate cu DAPI. Raportul N/C al imunoreactivității FUS a fost măsurat așa cum este descris în Fig. 1 B. Doar neuronii motori cu un nucleu vizibil au fost luați în considerare pentru analiză. Am folosit R și lme4 (Bates și colab., 2015) pentru a efectua o analiză liniară a efectelor mixte a relației dintre localizarea FUS și boala sALS. Bară de scară = 20 μm. Datele prezentate sunt raportul N/C (medie ± SD) per celulă din cel puțin opt cazuri pe grup. (B) Zonele nucleare și citoplasmatice ale nucleilor neuronilor motori și citoplasmei în sALS (n = 12) versus control (n = 8). Datele prezentate sunt medii ± SD. Valorile P din testul non-parametric al lui Mann-Whitney. (C) Măsurarea cu o singură celulă a zonei nucleare (dreapta) și citoplasmatică (stânga) nu prezintă nicio corelație cu raportul FUS nuclear-citoplasmatic, așa cum se arată în graficele dispersate. PCC = coeficient de corelație Pearson.
O reducere a dimensiunii soma neuronală motorie a fost prezentată anterior într-un model in vitro al SLA care se asociază cu apoptoza crescută (Kiskinis și colab., 2014). Am investigat astfel dacă modificările în localizarea subcelulară FUS în probe sporadice de SLA însoțesc modificări ale morfologiei celulare. Folosind analiza cu o singură celulă a dimensiunii nucleare și a celulelor, am demonstrat reduceri semnificative atât în zonele nucleare, cât și în cele citoplasmatice în SLA sporadică versus neuronii motori de control (Fig. 2 B), care nu s-au corelat cu reducerea localizării nucleare la citoplasmatice a FUS în sALS cazuri (Fig. 2 C).
FUS se leagă la un intron reținut aberant în transcrierea SFPQ, care este exportată din nucleu
După ce am găsit dovezi clare că mislocalizarea nucleară către citoplasmatică a FUS este mai răspândită în SLA decât s-a recunoscut anterior, am căutat să înțelegem interacțiunea moleculară cu 167 transcrieri aberante de păstrare a intronului pe care le-am descris recent în SLA (Luisier și colab., 2018). În acest scop am analizat datele iCLIP, care au permis identificarea țintelor de legare a ARN ale proteinei FUS. Folosind această abordare, am constatat că proteina FUS se leagă extensiv la intronul reținut aberant 9 în transcrierea SFPQ (Fig. 3 A și B), pe care l-am identificat ca fiind cel mai semnificativ intron reținut între diverse mutații ALS (Luisier și colab., 2018 ).). Am confirmat apoi că transcriptul de păstrare a intronului SFPQ este exportat în citoplasmă folosind fracționarea celulară citoplasmatică nucleară și qPCR (Fig. 3 C), arătând o proporție crescută de transcript de păstrare a intronului SFPQ în citosolul culturilor mutante VCP. Am folosit o hibridizare in situ a fluorescenței ARN cu o singură moleculă (smFISH) ca validare ortogonală că acest transcript este exportat din nucleu (Fig. 3 D).
Discuţie
Rezultatul principal al studiului nostru este că proteina FUS este localizată greșit de la nucleu la citoplasmă în cazuri dincolo de SLA legată de mutația FUS. Mai exact, găsim o localizare incorectă a FUS în SLA legată de mutația VCP și, în mod crucial, în SLA sporadică. Descoperirile noastre au fost atent validate încrucișat în liniile iPSC (patru mutante VCP și patru linii iPSC de control), un model transgenic de șoarece (trei mutante VCP și trei șoareci de control) și țesut post-mortem (12 cazuri ALS sporadice și opt cazuri de control) ). Mislocalizarea omniprezentă a FUS a evitat probabil detectarea până în momentul în care FUS rămâne în mare parte neagregată în citoplasmă, mai degrabă decât să facă parte din agregatele TDP-43 în cazuri sporadice de SLA.
Descoperirile noastre susțin un model prin care mislocalizarea FUS de la nucleu la citoplasmă are loc în majoritatea cazurilor de SLA, dar în general nu pare să se agregeze în citoplasmă. Pierderea nucleară a proteinei FUS poate afecta splicarea pre-ARNm, în timp ce posibilitatea unui câștig toxic citosolic al funcției este, de asemenea, de remarcat în lumina studiilor recente (López-Erauskin și colab., 2018). Interesant, agregarea TDP-43 este observată și în majoritatea cazurilor SLA familiale și sporadice, cu excepția celor cauzate de mutațiile SOD1. Astfel, localizarea incorectă a FUS pare să apară într-o majoritate similară a cazurilor cu agregarea TDP-43. Important, FUS localizat greșit este observat la modelele de șoarece și iPSC în stadii incipiente, înainte de apariția agregatelor TDP-43, indicând faptul că FUS neagregat mal localizat ar putea juca un rol cauzal la începutul procesului bolii SLA, probabil prin crearea unei agregări mai mari- mediu citoplasmatic predispus.
Analiza noastră a datelor iCLIP sugerează că FUS se leagă avid de intronul reținut aberant al transcrierii SFPQ în ALS. În mod cumulativ, datele noastre sunt în concordanță cu o ipoteză de lucru că FUS de tip sălbatic ar putea călători în afara nucleului atunci când este legat de intronul reținut aberant 9 al transcrierii SFPQ în ALS (Fig. 3 E), deși confirmarea unui astfel de mecanism va necesita lucrări detaliate de urmărire moleculară.
Pe scurt, raportăm o mislocalizare largă nerecunoscută anterior (dar nu și agregare) a FUS în SLA și propunem un mecanism specific contextual presupus pentru aceasta prin interacțiunea sa cu intronul 9 păstrat aberant în transcrierile SFPQ. Aceste constatări ridică perspectiva orientării către localizarea greșită nuclear-citoplasmatică a FUS neagregat ca strategie terapeutică presupusă în SLA.
- Ce este întrebările frecvente, tehnicile și rețetele despre gastronomia moleculară
- Beneficiile pentru sănătate din spatele jocului de golf - Golf Hallmark
- Baza moleculară a alergiilor la arahide
- Caviarul de pepene verde, o introducere în gastronomia moleculară, mâncare prăjită, fitness și tehnologie
- Metabolomica ne țintită relevă efectele moleculare ale dietei ketogene asupra xenogrefei sănătoase și tumorale